تقنية النانو للدنا
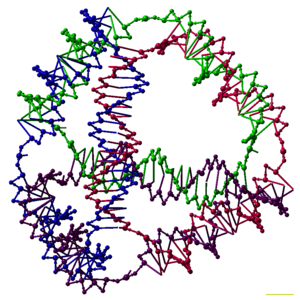
تقنية الدنا النانوية هي أحد فروع تقانة الصغائر التي تعتمد على مجموعة سمات التعرف الجزيئي إنگليزية: molecular recognition للحمض النووي (دنا) بالإضافة إلى باقي الأحماض النووية بهدف تصنيع هياكلٍ اصطناعيةٍ مصممةٍ من الدنا ليتم استخدامها بعد ذلك في الأغراض التقنية المختلفة. مما يدعونا إلى معرفة أن الدنا يُستَخْدَمُ هنا كمادةٍ بنائيةٍ بدلاً من كونه مجرد حاملٍ وناقلٍ للمعلومات الوراثية، مما يجعل ذلك العلم أحد أمثلة علم الاحياء النانوي. ولتقانة الدنا الحيوية تطبيقاتٍ عدةٍ في مجال التجميع الذاتي الجزيئي وحوسبة الدنا.
فعلى الرغم من أن الدنا غالباً ما يُعتبر ناقل وحامل المعلومات الوراثية في الخلايا الحية في إطار علم الأحياء الجزيئي، فإن تقانة الدنا الجزيئية تهتم بالدنا فردياً كمادةٍ وكمركبٍ كيميائيٍ، وغالباً ما تتبعه خارج أي إطارٍ حيويٍ. ومن ثم، فتقانة الدنا الجزيئية تستفيد من حقيقة أنه بسبب خصوصية زوج واتسون كريك القاعدي، فإن أجزاءً وقطعاً فقط من ضفائر الدنا، والتي تُعَدُ تكميليةً لكلٍ منها الآخر، سترتبط بكلٍ منها الأخرى بهدف تشكيل حلزون الدنا المزدوج إنگليزية: Nucleic acid double helix. هذا ويحاول علم تقانة الدنا النانوي تصميم نسبياً إنگليزية: Nucleic acid design ضفائر الدنا ومن ثم فالقطع المرغوبة فقط من كل ضفيرةٍ ستتجمع في الأماكن الصحيحة لتكوين هيكلاً مستهدفاً مرغوباً.
هذا ويبد أن هذا المجال يُطلق عليه اسم (تقانة الدنا النانوية)، إلا أن مبادئه تنطبق كذلك وبصورةٍ متكافئةٍ على الأحماض النووية الأخرى والتي منها الحمض الريبي النووي وحمض الببتيد النووي إنگليزية: Peptide Nucleic Acid، هذا بالإضافة إلى أنه تم إنتاج هياكلٍ وبناياتٍ تدمجهم معاً. مما دعى إلى الإشارة إلى هذا المجال على أنه تقانة الحمض النووي النانوية جراء ذلك السبب.
مفاهيم أساسية
خصائص الأحماض النووية
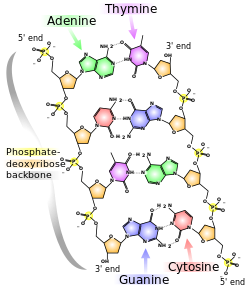
يقوم مجال تقانة الدنا النانوية بإنتاج هياكلٍ مركبةٍ من الأحماض النووية من خلال الاستفادة من خصوصية الزوج القاعدي لجزيئات الحمض النووي. وتتكون بنية جزيء الحمض النووي من سلسلةٍ من النيكلوتيدات، المتميزة بالقواعد النووية التي تحتوي عليها. حيث تكون الأربع قواعدٍ النيتروجينيةٍ المستخدمة في حمض الدنا النووي الأدينين A، الگوانين G، الثيمين C والسيتوزين T. كما أن للحمض النووي خاصية أن كل جزيئين إثنين يرتبطان بكلٍ منهما الآخر لتشكيل حلزونٍ مزدوجٍ إنگليزية: double helix، وذلك فقط في حالة كون هاتين السلسلتين متكاملتين إنگليزية: Complementarity (molecular biology)، مما يعني أنهما تشكلان سلاسلاً متطابقةً من الأزواج القاعدية، مع ارتباط قواعد الأدينين فقط إلى قواعد السيتوزين وقواعد الثيمين إلى قواعد الگوانين. وبسبب أن تكون أزواج قاعدية متطابقة تماماً مواتيةٌ بقوةٍ إنگليزية: Nucleic acid thermodynamics، فمن المتوقع أن ترتبط ضفائر الحمض النووي ببعضها الآخر في التعديل الخاص بتضخيم عدد الأزواج القاعدية الصحيحة. كما تُستخدم تلك الخاصية، الخاصة بكون تلك السلسلة تحدد شكل الرابطة والبنية الكاملة، في تطبيقات تقانة الدنا النانوية بمثل تلك التسلسلات المصممة نسبياً إنگليزية: Nucleic acid design، مما يسفر عن القابلية لتشكيل بنيةٍ مرغوبةٍ.
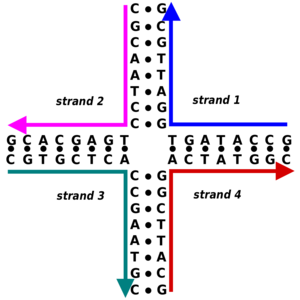
تستخدم كل هياكل تقانة الدنا النانوية تقريباً هياكل الدنا المتفرعة المحتوية على تقاطعاتٍ، وذلك تعارضاً مع غالبية الدنا الحيوي المتواجد في صيغة حلزون الدنا المزدوج إنگليزية: Nucleic acid double helix الخطية. وهنا نلاحظ أن واحداً من أبسط الهياكل المتفرعة، والتي صُنِعَت أولاً، هو تقاطع رباعي الأذرع والذي يمكن صناعته باستخدام ضفائر الحمض النووي (دنا) الفردية والتي تتسم بأنها متكاملة مع بعضها الآخر في النموذج الصحيح. هذا وعلى خلاف ما هو قائمٌ في حالة تقاطعات هوليداي إنگليزية: Holliday junction، فإن التسلسل القاعدي إنگليزية: Nucleic acid sequence في كل ذراعٍ، في حالة التقاطع رباعي الأذرع الصناعي غير المتحرك والموضح بالأسفل، يكون مختلفاً عن الآخر، مما يعني أن نقطة هذا التقاطع ثابتةٌ في موقعٍ محددٍ.
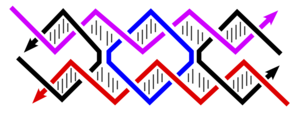
هذا ويمكن استخدام التقاطعات في الجزيئات الأكثر تعقيداً. حيث أن واحداً من أكثر تلك الجزيئات واسعة الاستخدام يتمثل في "التقاطع المزدوج" أو حافز دي إكس (DX). فيمكن تخيل جزيء دي إكس (DX) على أنه مكون من حلزونين إثنين من الدنا، المتوازيين مع بعضهما الآخر، مع وجود نقطتين للتقاطع حيث تعبر الضفائر من حلزونٍ واحدٍ إصوب الآخر. ويلاحظ أن كل نقطة تقاطعٍ هي نفسها عبارة عن تقاطعٍ رباعي الأذرع طوبوغرافياً. ويتسم هذا الجزيء بميزة أن نقاط التقاطع يتم إعاقتها الآن صوب توجهٍ فرديٍ، حيث يتم معارضتها لتصبح مرنةً كما هو الحال في حالة التقاطع رباعي الأذرع. مما يجعل حافز دي إكس (جزيء دي إكس) ملائماً كحاجزٍ بنائيٍ تركيبيٍ لتركيبات الحمض النووي (دنا) الأضخم حجماً.[3]
المجالان الفرعيان
DNA nanotechnology is sometimes divided into two overlapping subfields: تقنية نانو الدنا البنيوية و تقنية نانو الدنا الديناميكية. تقنية نانو الدنا البنيوية، أحياناً تـُختصر SDN، يركز على تخليق وتمييز مجمعات الأحماض النووية والمواد التي تتجمع لتصبح حالة نهائية من التوازن الاستاتيكي. وفي الناحية الأخرى، فإن تقنية نانو الدنا الديناميكية تركز على المجمعات ذات السلوك غير المتزن المفيد مثل القدرة على reconfigure based on a chemical or physical stimulus. Some complexes, such as nucleic acid nanomechanical devices, combine features of both the structural and dynamic subfields.[4][5]
The complexes constructed in structural DNA nanotechnology use topologically branched nucleic acid structures containing junctions. (In contrast, most biological DNA exists as an unbranched double helix.) One of the simplest branched structures is a four-arm junction that consists of four individual DNA strands, portions of which are complementary in a specific pattern. Unlike in natural Holliday junctions, each arm in the artificial immobile four-arm junction has a different base sequence, causing the junction point to be fixed at a certain position. Multiple junctions can be combined in the same complex, such as in the widely used double-crossover (DX) structural motif, which contains two parallel double helical domains with individual strands crossing between the domains at two crossover points. Each crossover point is, topologically, a four-arm junction, but is constrained to one orientation, in contrast to the flexible single four-arm junction, providing a rigidity that makes the DX motif suitable as a structural building block for larger DNA complexes.[3][6]
Dynamic DNA nanotechnology uses a mechanism called toehold-mediated strand displacement to allow the nucleic acid complexes to reconfigure in response to the addition of a new nucleic acid strand. In this reaction, the incoming strand binds to a single-stranded toehold region of a double-stranded complex, and then displaces one of the strands bound in the original complex through a branch migration process. The overall effect is that one of the strands in the complex is replaced with another one.[4] In addition, reconfigurable structures and devices can be made using functional nucleic acids such as deoxyribozymes and ribozymes, which can perform chemical reactions, and aptamers, which can bind to specific proteins or small molecules.[7]
تقنية نانو الدنا البنيوية
تقنية نانو الدنا البنيوية، أحياناً تُختصر SDN، تركز على synthesizing and characterizing nucleic acid complexes and materials where the assembly has a static, equilibrium endpoint. The nucleic acid double helix has a robust, defined three-dimensional geometry that makes it possible to predict and design the structures of more complicated nucleic acid complexes. Many such structures have been created, including two- and three-dimensional structures, and periodic, aperiodic, and discrete structures.[5]
الأعقاد الممتدة
![The assembly of a DX array. Left, schematic diagram. Each bar represents a double-helical domain of DNA, with the shapes representing complementary sticky ends. The DX complex at top will combine with other DX complexes into the two-dimensional array shown at bottom.[2] Right, an atomic force microscopy image of the assembled array. The individual DX tiles are clearly visible within the assembled structure. The field is 150 nm across.](/w/images/thumb/f/fc/Mao-DXarray-schematic-small.gif/175px-Mao-DXarray-schematic-small.gif)
|
![The assembly of a DX array. Left, schematic diagram. Each bar represents a double-helical domain of DNA, with the shapes representing complementary sticky ends. The DX complex at top will combine with other DX complexes into the two-dimensional array shown at bottom.[2] Right, an atomic force microscopy image of the assembled array. The individual DX tiles are clearly visible within the assembled structure. The field is 150 nm across.](/w/images/thumb/d/d5/DX_DNA_array.png/175px-DX_DNA_array.png)
| |
The assembly of a DX array. Left, schematic diagram. Each bar represents a double-helical domain of DNA, with the shapes representing complementary sticky ends. The DX complex at top will combine with other DX complexes into the two-dimensional array shown at bottom.[2] Right, an atomic force microscopy image of the assembled array. The individual DX tiles are clearly visible within the assembled structure. The field is 150 nm across.
| ||
![An example of an aperiodic two-dimensional lattice that assembles into a fractal pattern. Left, the Sierpinski gasket fractal. Right, DNA arrays that display a representation of the Sierpinski gasket on their surfaces[10]](/w/images/thumb/8/80/SierpinskiTriangle.svg/175px-SierpinskiTriangle.svg.png)
|
![An example of an aperiodic two-dimensional lattice that assembles into a fractal pattern. Left, the Sierpinski gasket fractal. Right, DNA arrays that display a representation of the Sierpinski gasket on their surfaces[10]](/w/images/thumb/f/fb/Rothemund-DNA-SierpinskiGasket.jpg/175px-Rothemund-DNA-SierpinskiGasket.jpg)
| |
An example of an aperiodic two-dimensional lattice that assembles into a fractal pattern. Left, the Sierpinski gasket fractal. Right, DNA arrays that display a representation of the Sierpinski gasket on their surfaces[10]
| ||
البنى المنفصلة
التجميع المقولب
تقنية نانو الدنا الديناميكية
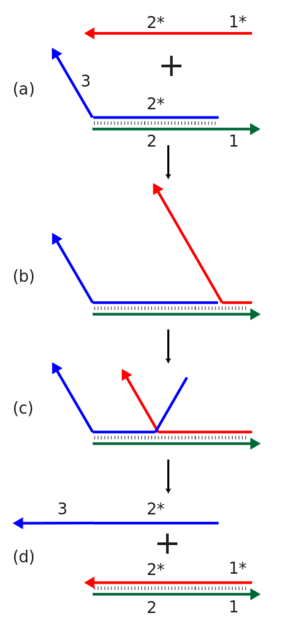
Strand displacement cascades
التصميم
يجب أن يتم تصميم هياكل الدنا النانوية حتى يتسنى لها أن تتجمع في الهياكل المرغوبة. ويتضمن هذا كلاً من تصميم هياكل الحمض النووي الثانوية إنگليزية: Nucleic acid secondary structure، الخاص بتقرير وتحديد أي الأجزاء التي منها يجب أن يتم ربط جزيئات الحمض النووي ببعضها البعض، وكذلك هياكل الحمض النووي الأولية إنگليزية: Nucleic acid primary structure، والخاص بتحديد هوية كل قاعدةٍ مفردةٍ.
التصميم التركيبي
لعل أول خطوةٍ في تصميم هياكل الحمض النووي النانوية تتمثل في تحديد كيفية تمثيل هيكلٍ متاحٍ بواسطة ترتيبٍ محددٍ لضفائر الحمض النووي. ومن ثم فخطوة التصميم تلك تحدد التركيب الثانوي لمركب الحمض النووي الذي سيقوم لاحقاً بالتجمع ضمن الشكل المرغوب. وهنا نلاحظ وجود العديد من المداخل التي تم توضيحها والمتمثلة فيما يلي:
- تقلص التماثل المتسلسل. حيث يركز غالبية التصميم في تقانة الدنا النانوية على تصميم سلاسل ومن ثم تكون البنية أو الهيكل المرغوب الوصول إليه هو عبارةٍ عن تدنٍ ديناميكيٍ حراريٍ، وتكون الهياكل التي أُسيء تجمعها ذات طاقاتٍ أعلى ومن ثم تكون غير مرغوبةٍ.
- الهياكل الملفوفة. لعل أحد المداخل البديلة للمنهجية البلاطية يتمثل في أن هياكل الدنا ثنائية الأبعاد يمكن إنتاجها من خلال ضفيرةٍ طويلةٍ مفردةٍ من التسلسل الجبري الذي يتم طيه أو لفه في الشكل المرجو بواسطة استخدام ضفائرٍ "مشبكيةٍ" أقصرٍ طولاً. مما يسمح بعد ذلك بإنتاج أشكالٍ ثنائية الأبعاد نانوية المقياس من خلال استخدام حمض الدنا النووي. وتضمنت التصاميم الموضحة الوجه المبتسم (سميلي) وخريطة شمال أمريكا التضاريسية غير المستوية. فقد كانت أوريگامي الدنا إنگليزية: DNA origami قصة غلاف الدورية العلمية نتيتشر في عددها الصادر في 15 من مارس 2006 م. [11]
- التجمع الحركي. ظهر اهتمام مؤخراً صوب ضبط حركة التجمع الذاتي للدنا، ومن ثم يمكن برمجة الديناميات العابرة إنگليزية: transient dynamics ضمن هذا التجمع. ونلاحظ أن لتلك الطريقة ميزةً تتمثل في التقدم بشكلٍ متساو الحرارة ومن ثم لا تتطلب خطوة التخمير إنگليزية: Annealing (biology) الحراري المطلوب في الطرق الديناميكية الحرارية الفردية. [12]
التصميم المتسلسل
 مقالة مفصلة: تصميم حمض نووي
مقالة مفصلة: تصميم حمض نووي
إنگليزية: Nucleic acid design
بعد استخدام وتطبيق أيٍ من الأساليب آنفة الذكر لتصميم الهياكل الثانوية للجزيء المستهدف، يجب تقسيم تسلسلٍ فعليٍ من النوكليوتيدات والتي ستتشكل في الهيكل المرغوب. وهنا يمثل تصميم الحمض النووي عملية إنتاج مجموعةٍ من سلاسل الأحماض النووية القاعدية والتي سترتبط ضمن تعديلٍ مرغوبٍ (انظر، على سبيل المثال، تركيب الحمض النووي إنگليزية: Nucleic acid structure). مما يجعل من تصميم الحمض النووي مركزياً في مجال تاقنة الدنا النانوية.
لتصميم الحمض النووي أهدافاً مثيلةً بتصميم البروتين إنگليزية: Protein design: ففي كليهما، يتم تصميم تسلسل المونومرات لصالح الهيكل المترابط أو المطوي الملفوف ولغير صالح الهياكل البديلة. وهنا نلاحظ أن لتصميم الحمض النووي ميزة كونه يمثل مشكلةً أبسط حسابياً، وذلك بسبب أن بساطة قواعد زوج واتسون كريك القاعدي تؤدي إلى سبلٍ حدسيةٍ بسيطةٍ والتي تسفر عن تصاميمٍ قويةٍ تجريبياً. على الرغم من ذلك، فإن هياكل الحمض النووي أقل تنوعاً من البروتينات في وظيفيتها. [13][14]
المواد والطرق
أنماط الهياكل
تم تصنيع وتمييز العديد من الهياكل المصنعة من الدنا.
الشبكات المتكررة (Periodic lattices)
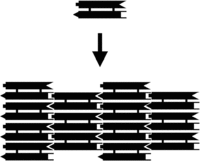

لعل إحدى الطرق الأولى لإنتاج هياكل الدنا النانوية تمثلت في تصنيعها من الوحدات المنفصلة الأصغر حجماً. ولتلك الطريقة ميزة كونها قادرةً على فصل التفاعلات الأقوى إدراكياً والتي تشكل كل بنيةٍ من تجمع الهيكل الكامل الأكبر حجماً. حيث أنها غالباً ما تُستخدم لإنتاج الشبكات الدورية، إلا أنه يمكن استخدامها كذلك لتحقيق التجمع الذاتي الحسابي إنگليزية: algorithmic self-assembly، مما يجعلها رصيفاً واحداً لحوسبة الدنا.
هذا بالإضافة إلى أنه يمكن تزويد وتجهيز جزيئات دي إكس أو ثنائية التقاطع بالنهايات اللزجة إنگليزية: sticky end بهدف دمجها ضمن الشبكة الدورية ثنائية الأبعاد. وهنا يكون لكل جزيء دي إكس أربعة أطرافٍ، واحد في كل نهايةٍ من النطاقين الحلزونيين المزدوجين الإثنين، وأن هذه يمكن تزويدها بنهاياتٍ لزجةٍ والتي تبرمجها ليتم دمجها ضمن نموذجٍ معينٍ. وهنا نلاحظ وجود أكثر من نمطٍ واحدٍ للجزيئات ثنائية التقاطع (دي إكس) يمكن استخدامها والتي يمكن إنتاجها ليتم ترتيبها في صفوفٍ أو أي نموذج فسيفساءٍ آخرٍ. ومن ثم فهي تشكل صفائحاً مسطحةً ممتدةً والتي هي عبارةٌ عن بلوراتٍ دنويةٍ ثنائية الأبعاد بصورةٍ أساسيةٍ. [16][17]
كما تم تشكيل المصفوفات ثنائية الأبعاد من المحفزات الأخرى كذلك، والتي منها مصفوفة تقاطع هوليداي معينة الأضلاع إنگليزية: Holliday junction rhombus array، وكذلك باقي المصفوفات ثنائية التقاطع (دي إكس) الأخرى العديدة والمتنوعة في أشكال المثلثات ومسدسات الأضلاع. [18][19][20]
ونلاحظ أن إنتاج الشبكات ثلاثية الأبعاد المصنعة من (الدنا) كان أولى الأهداف الخاصة بتقانة الدنا الحيوية، إلا أنه أثبت أنه واحداً من أصعب الأهداف ليتم تحقيقه. إلا أنه أفادت التقارير عام 2009 أنه تم النجاح في محاولات إنتاج شبكات الدنا ثلاثية الأبعاد، من خلال استخدام محفز قائم على فكرة الانشدادية إنگليزية: tensegrity، المتمثلة في التوازن القائم بين قوى التوتر والضغط. [21]
الأنابيب النانوية
بالإضافة إلى الصفائح المسطحة، تم إنتاج الشبكات ثنائية التقاطع (دي إكس) لتشكيل أنابيبٍ نانويةٍ جوفاء يتراوح قطرها من 4 إلى 20 نانومتراً. وهنا نلاحظ أن أنابيب الدنا النانوية تلك شبيهةٌ إلى حدٍ ما في أحجامها بالأنابيب النانوية الكربونية، إلا أن الأنابيب النانوية الكربونية عبارةٌ عن موصلاتٍ أقوى وأفضل للحرارة، في حين أنابيب الدنا النانوية هي أكثر قابليةٍ للتعديل بسهولةٍ وارتباطاً بالهياكل الأخرى. [22]
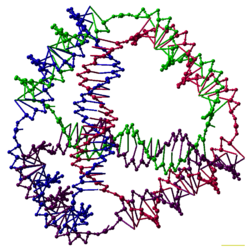
متعددة الأوجه
تم إنتاج عدداً من جزيئات الدنا ثلاثية الأبعاد والتي تتسم بالقدرة على الارتباط بمتعدد الأسطح والتي منها على سبيل المثال ثماني السطوح أو المكعب. وبصيغةٍ أخرى، فإن ثنائيات الدنا تتبع حواف متعددات الأسطح ذات تقاطع الدنا في كل رأسٍ له.
فقد تضمنت التوضيحات الأولى لمتعدد أسطح الدنا كلاً من روابط الدنا إنگليزية: DNA ligase المتعددة وخطوات تصنيع المرحلة الصلبة إنگليزية: solid-phase synthesis بهدف إنتاج متعدد السطوح. [23] إلا أن الأعمال الحديثة أسفرت عن إنتاج متعدداً للأسطح والذي يتسم تصنيعه بالسهولة. وهذا يتضمن ثماني سطوح الدنا المصنوع من ضفيرةٍ فرديةٍ طويلةٍ مصممة لتنطوي داخل التعديل الصحيح، بالإضافة إلى رباعي السطوح الذي يمكن إنتاجه من أربعة ضفائرٍ للدنا في خطوةٍ واحدةٍ. [1][24]
أشكال تعسفية
بالإضافة إلى ما سبق ذكره، تم تصنيع كذلك هياكل دنا ذات أوجهٍ صلدةٍ، بواسطة استخدام طريقة أوريگامي الدنا إنگليزية: DNA origami. حيث يمكن برمجة مثل تلك الهياكل لتفتح وتُطْلِقُ حمولتها استجابةً لمثيرٍ أو تحفيزٍ معينٍ، مما يجعلها مفيدةً كأقفاصٍ جزيئيةٍ إنگليزية: Molecular encapsulation مبَرْمَجةٍ. [25][26]
هياكل الحمض النووي النانوية الوظيفية
تركز تقانة الدنا النانوية على إنتاج جزيئاتٍ ذات وظيفيةٍ مصممةٍ وهياكلٍ كذلك. حيث تم استعراض وتوضيح العديد من تصنيفات الأنظمة الوظيفية.
عمارة نانوية
كان أول من اقترح فكرة استخدام مصفوفات الدنا لقولبة تجمع الجزيئات الوظيفية الأخرى هو نادرين سيمان في عام 1987، [27] إلا أنه تم تحقيق التقدم مؤخراً فقط في تقليل أنواع تلك المشاريع للمارسة. ففي عام 2006، قام الباحثون بربط جسيمات الذهب النانوية إنگليزية: Gold nanoparticle تساهمياً بالبلاطة ثنائة التقاطع للدنا إنگليزية: DX-based tile وأظهروا أن التجمع الذاتي لهياكل الدنا قامت كذلك بتجميع الجزيئات النانوية التي تم إضافتها لهم. كما ظهر مشروع استضافة غير تساهمية في عام 2007، بواسطة استخدام متعددات أميد بيتر ديرفان إنگليزية: Peter B. Dervan على مصفوفة ثنائية التقاطع لترتيب بروتينات الاستريبتافيدين إنگليزية: Streptavidin على أنواعٍ خاصةٍ من البلاطات على مصفوفة الدنا. [28][29]
هذا في عام 2006، قام كلٌ من دوير ولابين باستعراض الأحرف "D" "N" و"A" المنتجة على مصفوفة 4x4 ثنائية التقاطع (دي إكس) بواسطة استخدام بروتين الاستريبتافيدين. [30] بينما تم استعراض في عام 2007 تجمعٍ هرميٍ قائمٍ على هذا المُدْخَل والذي يوضح المقاييس للمصفوفات الأكبر حجماً (8x8 و8.96 MD). [30]
كما ظهر اهتمام في استخدام تقانة الدنا النانوية لتجميع الأجهزة الإلكترونيات الجزيئية. ووصلاً لتلك الغاية، تم استخدام حمض الدنا النووي لتجميع الأنابيب النانوية الكربونينة أحادية الجدار ضمن مقاحل التأثير الحقلي. [30]
التجمع الذاتي الحسابي

انظر أيضاً: حوسبة الدنا
تم تطبيق تقانة الدنا الجزيئية في المجال المرتبط بها الخاص بحوسبة الدنا. حيث أنه قد يكون للبلاطات الدنوية ثنائية التقاطع (دي إكس) تسلسلات نهاياتها اللزجة المختارة ومن ثم فهي تسلك على أنها بلاطات وانج إنگليزية: Wang tiles، مما يسمح لهم بأداء الحسابات. كما تم استعراض المصفوفة ثنائية التقاطع (دي إكس) والتي يرمز لتجمعها بعملية الفصل الحصري إنگليزية: Exclusive or أو (XOR)؛ مما يسمح لمصفوفة الدنا بتنفيذ الخلايا ذاتية السلوك والتي تولِّد كسيريات يُطلق عليها اسم مثلث سيربنسكي. وهذا يوضح أن الحساب يمكن دمجه ضمن تجمعٍ من مصفوفات الدنا، مما يزيد مجال فيما وراء المصفوفات المتكررة البسيطة.
ولنلاحظ أن حوسبة الدنا تتداخل مع، ولكن ليست منفصلة عن، تقانة الدنا النانوية. حيث تستخدم الثانية خصوصية زوج واتسون- كريك القاعدي لإنتاج هياكلٍ جديدةٍ من الدنا. ويمكن استخدام هذه الهياكل المنتجة في مجال حوسبة الدنا، إلاأنها ليست ملزمة ليتم استخدامها لهذا الغرض. هذا بالإضافة إلى أن حوسبة الدنا يمكن إجراؤها بدون استخدام أنماط الجزيئات المنتجة بواسطة استخدام تقانة الدنا النانوية. [10]
أجهزة الدنا النانوميكانيكية
 مقالة مفصلة: آلة الحمض النووي
مقالة مفصلة: آلة الحمض النووي
تم إنتاج مركبات الدنا التي تغير واجهتها بناءً على بعض المثيرات. حيث يتمثل الغرض من تصنيعها في أن يكون لها تطبيقاتٍ في مجال روبوات النانو. حيث أُطلق على واحداً من أوائل تلك الأجهزة: "ملاقيط جزيئية"، والذي يقوم بالتغير من الوضع المفتوح إلى الوضع المغلق بناءً على وجود ضفائر التحكم. [31]
كما تم تصنيع آلات الدنا لتظهر حركةً لفافةً ملتويةً. كما أن أحد تلك الأجهزة يستفيد كذلك من الانتقال بين أشكال B-DNA إنگليزية: Nucleic acid double helix وZ-DNA بهدف الاستجابة للتغير في شروط الصد. [32] في حين تعتمد إحداها الأخرى على وجود ضفائر ضبطٍ للتحول من تشكيل تقاطع محاذاةٍ (PX) إنگليزية: Paranemic crossover إلى تشكيل تقاطعٍ مزدوجٍ (JX2). [33]
المواد والطرق

تتوفر بالفعل سلاسل الدنا التقليدية عبر عملية تصنيع قليل النيوكليوتيد إنگليزية: oligonucleotide synthesis. حيث غالباً ما تدار تلك العملية بواسطة استخدام آلة تصنيع الدنا، كما أن الدنا التقليدي أصبح متاحاً للتبادل التجاري لدى العديد من البائعين.
كما أن سلاسل ضفائر الدنا الفردية والتي تنتج الهياكل المستهدفة يتم تصميمها حوسبياً. هذا وتُستَخْدَم النموجة الجزيئية (Molecular modeling) والنمذجة الحرارية الديناميكية في بعض الأحيان كذلك لتحسين سلاسل الحمض النووي إلأى أحسن الأوضاع المرغوبة.
وتتسم جزيئات الدنا التي تم إنتاجها بواسطة استخدام تقانة الدنا الجزيئية غالباً بسمة الفصل الكهربائي للهلام، والتي توفر معلوماتٍ حول حجم وشكل جزيئات الدنا، مما يشير إلى ما إذا كان قد تم إنتاجها بصورةٍ ملائمةٍ كما هو مرغوبٍ أم لا. وكذلك يمكن استخدام كلٍ من الوسم الفلوري (Fluorescent labeling) ونقل طاقة رنين فورستر (Förster resonance energy transfer) بهدف تشخيص بنية الجزيئات.
هذا ويمكن تصوير هياكل الدنا مباشرةً بواسطة استخدام مجهر الطاقة الذرية، والذي يقوم بتصوير الهياكل المتواجدة على سطحٍ مسطحٍ مستوٍ. ونلاحظ أن تلك الطريقة تناسب الهياكل ثنائية الأبعاد بصورةٍ جيدةٍ، إلا أنها أقل فائدةٍ في حالة الهياكل ثلاثية الأبعاد المتفردة. حيث يصبح المجهر الإلكتروني النافذ (Transmission electron microscopy) والمجهر الإلكتروني شديد التبريد (cryo-electron microscopy) سبلاً ضروريةً هنا. ويتم تحليل المشابك ثلاثية الأبعاد الممتدة بواسطة استخدام دراسة البلورات بالأشعة السينية. في حين يمكن دراسة حركية تجمع الدنا الذاتي بواسطة تقنيات وأساليب الوقت الحقيقي والتي منها مثلاً التداخل ثنائي الاستقطاب إنگليزية: Dual polarization interferometry وQCMD.
التاريخ

كان أول من ابتكر فكرة تقانة الدنا الحيوية هو نادرين سيمان في أوائل الثمانينات من القرن العشرين. [34] حيث اهتم سيمان مبدئياً باستخدام ملقاط الدنا ثلاثي الأبعاد لتوجيه الجزيئات المستهدفة، والتي ستتحدد دراستها البلورية من خلال التخلص من العملية الصعبة الخاصة بالحصول على بلوراتٍ نقيةٍ. وأفادت التقارير أن تلك الفكرة كانت قد واتته في خريف 1980، بعد إدراكه التشابه فيما بين لوحة القطع الخشبي (ديبث) أو العمق لإشر ومصفوفة تقاطعات الدنا سداسية الأذرع. [3][35] ولتحقيق تلك الغاية، نشر معمل سيمان في عام 1991 عملية تصنيع مكعبٍ مصنوعٍ من الدنا، والذي يعتبر أول كائنٍ نانوي المقياس ثلاثي الأبعاد، والذي على أثره حصل سيمان على جائزة فينمان في مجال القتانة النانوية (Foresight Nanotech Institute Feynman Prize)، والذي كان قد تبعه تصنيع مجسم الدنا الثماني المبتور (truncated octahedron). على الرغم من ذلك، فقد أصبح من الواضح لاحقاً أن هذه الجزيئات، الأشكال متعددة الأضلاع ذات التقاطعات المرنة كنقاطها الهندسية، لم تكن صلبةً بصورةٍ كافيةٍ لتشكيل الملاقيط ثلاثية الأبعاد الممتدة.[3][34]
في حين طور سيمان حافز إنگليزية: Structural motif التقاطع المزدوج الصلب، بالإضافة إلى أنه، وبالتعاون مع إريك وينفري (Erik Winfree)، نشر عام 1998 إنتاج الملاقيط ثنائية الأبعاد من البلاطات مزدوجة التقاطع (DX). حيث تتسم تلك الهياكل القائمة على استخدام البلاطات بأنها توفر القدرة على تطبيق وتنفيذ استخدام حوسبة الدنا، والتي أوضحها وينفري وبول روزاموند في عام 2004، والتي بفضلها اقتسما معاً جائزة فينمان للتقانة النانوية في عام 2006.[3][34]
ومع مرور الزمن، يستمر مجال تقانة الدنا النانوية في التشعب أكثر وأكثر. فأول آلةٍ دنا نانويةٍ - الحافز الذي يغير بنيته استجابةً لمدخلٍ ما - ظهرت عام 1999. كما كان سيمان أول من اقترح مجال العمارة النانوية في عام 1987، والتي بدأت في الظهور عام 2006. هذا وكان روزاموند أول من استعرض أسلوب أوريگامي الدنا في عام 2006 لتسهيل إنتاج جزيئات الدنا المطوية الملتفة لأي شكل. أما في عام 2009، نشر سيمان تصنيع ملقاطٍ ثلاثي الأبعاد، بعد مرور ما يقرب من 30 عاماً من استعداده للقيام بذلك.
انظر أيضا
المصادر
مراجع
- ^ أ ب ت DNA polyhedra: Goodman, R.P. (2005). "Rapid chiral assembly of rigid DNA building blocks for molecular nanofabrication". ساينس. 310 (5754): 1661–1665. doi:10.1126/science.1120367. ISSN 0036-8075. PMID 16339440.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help) - ^ أ ب ت ث Overview: Mao, Chengde (December 2004). "The emergence of complexity: lessons from DNA". PLoS Biology. 2 (12): 2036–2038. doi:10.1371/journal.pbio.0020431. PMC 535573. PMID 15597116.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ أ ب ت ث ج ح Overview: Seeman, Nadrian C. (2004). "Nanotechnology and the double helix". ساينتفك أمريكان. 290 (6): 64–75. doi:10.1038/scientificamerican0604-64. PMID 15195395.
{{cite journal}}: Unknown parameter|month=ignored (help) - ^ أ ب Dynamic DNA nanotechnology: Zhang, D. Y.; Seelig, G. (February 2011). "Dynamic DNA nanotechnology using strand-displacement reactions". Nature Chemistry. 3 (2): 103–113. Bibcode:2011NatCh...3..103Z. doi:10.1038/nchem.957. PMID 21258382.
- ^ أ ب Structural DNA nanotechnology: Seeman, Nadrian C. (November 2007). "An overview of structural DNA nanotechnology". Molecular Biotechnology. 37 (3): 246–257. doi:10.1007/s12033-007-0059-4. PMC 3479651. PMID 17952671.
- ^ خطأ استشهاد: وسم
<ref>غير صحيح؛ لا نص تم توفيره للمراجع المسماةSeeman2010 - ^ Dynamic DNA nanotechnology: Lu, Y.; Liu, J. (December 2006). "Functional DNA nanotechnology: Emerging applications of DNAzymes and aptamers". Current Opinion in Biotechnology. 17 (6): 580–588. doi:10.1016/j.copbio.2006.10.004. PMID 17056247.
- ^ Other arrays: Strong, Michael (March 2004). "Protein Nanomachines". PLoS Biology. 2 (3): e73. doi:10.1371/journal.pbio.0020073. PMC 368168. PMID 15024422.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Yan, H.; Park, S. H.; Finkelstein, G.; Reif, J. H.; Labean, T. H. (26 September 2003). "DNA-templated self-assembly of protein arrays and highly conductive nanowires". Science. 301 (5641): 1882–1884. Bibcode:2003Sci...301.1882Y. doi:10.1126/science.1089389. PMID 14512621.
- ^ أ ب ت Algorithmic self-assembly: Rothemund, Paul W. K. (2004). "Algorithmic Self-Assembly of DNA Sierpinski Triangles". PLoS Biology. 2 (12): 2041–2053. doi:10.1371/journal.pbio.0020424. ISSN 1544-9173. PMC 534809. PMID 15583715.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help)CS1 maint: unflagged free DOI (link) - ^ DNA origami: Rothemund, Paul W. K. (2006). "Folding DNA to create nanoscale shapes and patterns". Nature. 440 (7082): 297–302. doi:10.1038/nature04586. ISSN 0028-0836. PMID 16541064.
{{cite journal}}: Cite has empty unknown parameter:|month=(help) - ^ Kinetic assembly: Yin, Peng; Choi, Harry M. T.; Calvert, Colby R.; Pierce, Niles A. (2008). "Programming biomolecular self-assembly pathways". Nature. 451 (7176): 318–22. doi:10.1038/nature06451. PMID 18202654.
- ^ Sequence design: Dirks, Robert M. (2004). "Paradigms for computational nucleic acid design". Nucleic Acids Research. 32 (4): 1392–1403. doi:10.1093/nar/gkh291. PMC 390280. PMID 14990744.
{{cite journal}}: Cite has empty unknown parameter:|month=(help); Unknown parameter|coauthors=ignored (|author=suggested) (help) - ^ Sequence design: Dirks, Robert M.; Bois, Justin S.; Schaeffer, Joseph M.; Winfree, Erik; Pierce, Niles A. (2007). "Thermodynamic Analysis of Interacting Nucleic Acid Strands". SIAM Review. 49: 65. doi:10.1137/060651100.
- ^ Strong, Michael (2004). "Protein Nanomachines". PLoS Biology. 2 (3): e73. doi:10.1371/journal.pbio.0020073. PMC 368168. PMID 15024422.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ DX arrays: Winfree, Erik (1998). "Design and self-assembly of two-dimensional DNA crystals". Nature. 394 (6693): 529–544. doi:10.1038/28998. ISSN 0028-0836. PMID 9707114.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help) - ^ Liu, Furong (1999). "Modifying the Surface Features of Two-Dimensional DNA Crystals". Journal of the American Chemical Society. 121 (5): 917–922. doi:10.1021/ja982824a. ISSN 0002-7863.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help) - ^ Other arrays: Mao, Chengde (1999). "Designed Two-Dimensional DNA Holliday Junction Arrays Visualized by Atomic Force Microscopy". Journal of the American Chemical Society. 121 (23): 5437–5443. doi:10.1021/ja9900398. ISSN 0002-7863.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help) - ^ Other arrays: Constantinou, Pamela E. (2006). "Double cohesion in structural DNA nanotechnology". Organic and Biomolecular Chemistry. 4 (18): 3414–3419. doi:10.1039/b605212f. PMID 17036134.
{{cite journal}}: Cite has empty unknown parameter:|month=(help); Unknown parameter|coauthors=ignored (|author=suggested) (help) - ^ Other arrays: Mathieu, Frederick (2005). "Six-Helix Bundles Designed from DNA". Nano Letters. 5 (4): 661–665. doi:10.1021/nl050084f. ISSN 1530-6984. PMID 15826105.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help) - ^ Three-dimensional lattices: Zheng, Jianping; Birktoft, Jens J.; Chen, Yi; Wang, Tong; Sha, Ruojie; Constantinou, Pamela E.; Ginell, Stephan L.; Mao, Chengde; Seeman, Nadrian C. (2009). "From molecular to macroscopic via the rational design of a self-assembled 3D DNA crystal". Nature. 461 (7260): 74–7. doi:10.1038/nature08274. PMC 2764300. PMID 19727196.
- ^ DNA nanotubes: Rothemund, Paul W. K. (2004). "Design and Characterization of Programmable DNA Nanotubes". Journal of the American Chemical Society. 126 (50): 16344–16352. doi:10.1021/ja044319l. ISSN 0002-7863. PMID 15600335.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help) - ^ DNA polyhedra: Zhang, Yuwen (1994). "Construction of a DNA-truncated octahedron". Journal of the American Chemical Society. 116 (5): 1661–1669. doi:10.1021/ja00084a006. ISSN 0002-7863.
{{cite journal}}: Cite has empty unknown parameter:|month=(help); Unknown parameter|coauthors=ignored (|author=suggested) (help) - ^ DNA polyhedra: Shih, William M. (2004). "A 1.7-kilobase single-stranded DNA that folds into a nanoscale octahedron". [[نيتشر (مجلة)|]]. 427 (6975): 618–621. doi:10.1038/nature02307. ISSN 0028-0836. PMID 14961116.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help) - ^ DNA boxes: Andersen, Ebbe S.; Dong, Mingdong; Nielsen, Morten M.; Jahn, Kasper; Subramani, Ramesh; Mamdouh, Wael; Golas, Monika M.; Sander, Bjoern; Stark, Holger (2009). "Self-assembly of a nanoscale DNA box with a controllable lid". Nature. 459 (7243): 73–6. doi:10.1038/nature07971. PMID 19424153.
- ^ DNA boxes: Ke, Yonggang; Sharma, Jaswinder; Liu, Minghui; Jahn, Kasper; Liu, Yan; Yan, Hao (2009). "Scaffolded DNA Origami of a DNA Tetrahedron Molecular Container". Nano Letters. 9 (6): 2445–7. doi:10.1021/nl901165f. PMID 19419184.
- ^ Nanoarchitecture: Robinson, Bruche H. (1987). "The Design of a Biochip: A Self-Assembling Molecular-Scale Memory Device". Protein Engineering. 1 (4): 295–300. doi:10.1093/protein/1.4.295. ISSN 0269-2139. PMID 3508280.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help) - ^ Nanoarchitecture: Zheng, Jiwen (2006). "2D Nanoparticle Arrays Show the Organizational Power of Robust DNA Motifs". Nano Letters. 6 (7): 1502–1504. doi:10.1021/nl060994c. ISSN 1530-6984. PMID 16834438.
{{cite journal}}: Cite has empty unknown parameter:|month=(help); Unknown parameter|coauthors=ignored (|author=suggested) (help) - ^ Nanoarchitecture: Cohen, Justin D. (2007). "Addressing Single Molecules on DNA Nanostructures". Angewandte Chemie. 46 (42): 7956–7959. doi:10.1002/anie.200702767. ISSN 0570-0833. PMID 17763481.
{{cite journal}}: Cite has empty unknown parameter:|month=(help); Unknown parameter|coauthors=ignored (|author=suggested) (help) - ^ أ ب ت Park, Sung Ha (2006). "Finite-Size, Fully Addressable DNA Tile Lattices Formed by Hierarchical Assembly Procedures". Angewandte Chemie. 118 (40): 749–753. doi:10.1002/ange.200690141. ISSN 1521-3757.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help) - ^ DNA machines: Yurke, Bernard (2000). "A DNA-fuelled molecular machine made of DNA". Nature. 406 (6796): 605–609. doi:10.1038/35020524. ISSN 0028-0836. PMID 10949296.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help) - ^ DNA machines: Mao, Chengde (1999). "A DNA Nanomechanical Device Based on the B-Z Transition". Nature. 397 (6715): 144–146. doi:10.1038/16437. ISSN 0028-0836. PMID 9923675.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help) - ^ DNA machines: Yan, Hao (2002). "A robust DNA mechanical device controlled by hybridization topology". Nature. 415 (6867): 62–65. doi:10.1038/415062a. ISSN 0028-0836. PMID 11780115.
{{cite journal}}: Unknown parameter|coauthors=ignored (|author=suggested) (help); Unknown parameter|month=ignored (help) - ^ أ ب ت History: . New York.
{{cite book}}: Missing or empty|title=(help); Unknown parameter|الأخير=ignored (help); Unknown parameter|الأول=ignored (help); Unknown parameter|الرقم المعياري=ignored (help); Unknown parameter|الصفحات=ignored (help); Unknown parameter|العنوان=ignored (help); Unknown parameter|الناشر=ignored (help); Unknown parameter|سنة=ignored (help) - ^ History: See Nadrian Seeman's homepage, Current crystallization protocol for a statement of the problem, and Nadrian Seeman's homepage, DNA cages containing oriented guests for the proposed solution.
قراءات إضافية
تتبع قائمة المقالات والكتب التي تستطيع توفير مزيداً من المعلومات للقاريء في مجال تقانة الدنا النانوية:
- Seeman, Nadrian C. (2004). "التقانة النانوية والحلزون المزدوج". ساينتفك أمريكان. 290 (6): 64–75. doi:10.1038/scientificamerican0604-64. PMID 15195395.
{{cite journal}}: Unknown parameter|month=ignored (help)—مقالة كتبها مؤسس المجال للعاملين العاديين. - "Chapter 8: DNA Self-Assembly". New York.
{{cite book}}: Missing or empty|title=(help); Unknown parameter|الأخير=ignored (help); Unknown parameter|الأول=ignored (help); Unknown parameter|الرقم المعياري=ignored (help); Unknown parameter|العنوان=ignored (help); Unknown parameter|الناشر=ignored (help); Unknown parameter|سنة=ignored (help)—استعراض للنتائج الحقلية من منظور التجمع الذاتي. - Seeman, Nadrian C. (1999). "هندسة الدنا وتطبيقاتها في مجال التقانة النانوية". Trends in Biotechnology. 17 (11): 437–443. doi:10.1016/S0167-7799(99)01360-8. ISSN 0167-7799.
{{cite journal}}: Unknown parameter|month=ignored (help)—مقالة أقدم تحتوي على وصف جيد للافعية القائمة وراء تقانة الدنا. - Seeman, Nadrian C. (2007). "استعراض لتقانة الدنا النانوية البنائية". Molecular Biotechnology. 37 (3): 246–57. doi:10.1007/s12033-007-0059-4. PMID 17952671.—مقالة أكثر حداثة.
- Feldkamp, Udo; Niemeyer, Christof M. (2006). "التصميم النسبي لعمارة الدنا النانوية". Angewandte Chemie International Edition. 45 (12): 1856–76. doi:10.1002/anie.200502358. PMID 16470892.&mdashمقال جيد من وجهة نظر تصميم الهياكل الثانوية.
- Lin, Chenxiang; Liu, Yan; Rinker, Sherri; Yan, Hao (2006). "التجمع الذاتي القائم على بلاطة الدنا: بناء هياكل معمارية نانوية معقدة". ChemPhysChem. 7 (8): 1641–7. doi:10.1002/cphc.200600260. PMID 16832805.—مقال مصغر يدور أساساً على التركيز على التجمع القائم على التبلط.
- Chen, Junghuei; Jonoska, Natasha; Rozenberg, Grzegorz (eds.). Natural سلسلة الحوسبة. New York.
{{cite book}}: Missing or empty|title=(help); Unknown parameter|الرقم المعياري=ignored (help); Unknown parameter|العنوان=ignored (help); Unknown parameter|الناشر=ignored (help); Unknown parameter|سنة=ignored (help)—كتاب يشتمل على مقالاتٍ للعديد من الباحثين في مجال تقانة الدنا النانوية وحوسبة الدنا.
وصلات خارجية
- Chengde Mao page at جامعة پوردو [1]
- John Reif lab at جامعة ديوك [2]
- Nadrian Seeman lab at جامعة نيويورك [3]
- William M. Shih lab at مدرسة طب هارڤررد [4]
- Andrew Turberfield lab at جامعة أكسفورد [5]
- Erik Winfree lab at معهد كاليفورنيا للتكنولوجيا [6]
- Hao Yan lab at جامعة ولاية أريزونا [7]
- Bernard Yurke page at مختبرات بل [8]
- Software for DNA modeling - Ascalaph DNA
- Software for Structural DNA Nanotechnology (SDN) design - GIDEON
- International Society for Nanoscale Science, Computation and Engineering [9]

