QPCR
تفاعل الپوليمريز المتسلسل بالزمن الحقيقي ( PCR بالزمن الحقيقي ) ، والمعروف أيضًا باسم تفاعل الپوليمريز المتسلسل الكمي ( qPCR ) ، هو تقنية مختبرية في علم الأحياء الجزيئي تعتمد على تفاعل الپوليمريز المتسلسل (PCR). يراقب تضخيم جزيء الدنا المستهدف خلال تفاعل الپوليمريز المتسلسل (أي في الزمن الحقيقي) ، وليس في نهايته ، كما هو الحال في تفاعل الپوليمريز المتسلسل التقليدي. يمكن استخدام تفاعل الپوليمريز المتسلسل في الوقت الفعلي كمياً (تفاعل الپوليمريز المتسلسل الكمي بالزمن الحقيقي) وشبه الكمي (أي فوق / تحت كمية معينة من جزيئات الدنا) (تفاعل الپوليمريز المتسلسل شبه الكمي في الزمن الحقيقي).
هناك طريقتان شائعتان للكشف عن نواتج تفاعل الپوليمريز المتسلسل في تفاعل الپوليمريز المتسلسل بالزمن الحقيقي هي (1) أصباغ فلورية غير محددة تتقارب مع أي دنا مزدوج الشريط و (2) مسابير الدنا محددة التسلسل تتكون من قليل النوكليوتيدات الموصوفة بمراسل فلوري. ، الذي يسمح بالكشف فقط بعد تهجين المسبار بتسلسله التكميلي.
تقترح المعلومات الصغيرة لنشر التجارب الكمية بالزمن الحقيقي لتجارب تفاعل الپوليمريز المتسلسل (MIQE) استخدام الاختصار qPCR في تفاعل الپوليمريز المتسلسل الكمي بالزمن الحقيقي واستخدام RT-qPCR للنسخ العكسي - qPCR. .[1] يشير اختصار "RT-PCR" بشكل شائع إلى تفاعل الپوليمريز المتسلسل النسخ العكسي وليس تفاعل الپوليمريز المتسلسل بالزمن الحقيقي ، ولكن لا يلتزم جميع المؤلفين بهذه الاتفاقية.[2]
الخلفية
تنظم الخلايا في جميع الكائنات التعبير الجيني عن طريق دوران النسخ الجينية ( رنا وحيد الشريط): يمكن قياس كمية الجين المعبر عنه في الخلية بعدد نسخ نسخة الرنا لهذا الجين الموجود في العينة. من أجل الكشف عن التعبير الجيني وقياسه من كميات صغيرة من الرنا ، من الضروري تضخيم نسخة الجين. تفاعل الپوليمريز المتسلسل (PCR) هو طريقة شائعة لتضخيم الحمض النووي. بالنسبة لـتفاعل الپوليمريز المتسلسل القائم على الرنا ، يتم نسخ عينة الرنا أولاً إلى دنا تكميلي (cDNA) مع المنتسخة العكسية .
من أجل تضخيم كميات صغيرة من الدنا ، يتم استخدام نفس المنهجية كما هو الحال في تفاعل الپوليمريز المتسلسل التقليدي باستخدام قالب الدنا ، زوج واحد على الأقل من المشرعات المحددة ، ديوكسي ريبونيوكليوتيد ، محلول منظم مناسب وپوليمريز الدنا المستقر حراريًا . تتم إضافة مادة مميزة بفلوروفور إلى هذا الخليط في جهاز تدوير حراري يحتوي على أجهزة استشعار لقياس فلورية الفلوروفور بعد أن يتم استثارته على الطول الموجي المطلوب مما يسمح بقياس معدل التوليد لمنتج واحد أو أكثر من النواتج المحددة. هذا يسمح بقياس معدل توليد المنتج المكبر في كل دورة تفاعل الپوليمريز المتسلسل . يمكن تحليل البيانات الناتجة عن طريق برامج الكمبيوتر لحساب التعبير الجيني النسبي (أو رقم نسخة مرسال الحمض النووي الريبي ) في عدة عينات. يمكن أيضًا تطبيق تفاعل الپوليمريز المتسلسل الكمي للكشف عن الحمض النووي وتحديده في العينات لتحديد وجود ووفرة تسلسل معين من الحمض النووي في هذه العينات.[3] يتم إجراء هذا القياس بعد كل دورة تضخيم ، وهذا هو السبب في أن هذه الطريقة تسمى تفاعل الپوليمريز المتسلسل بالزمن الحقيقي (أي تفاعل الپوليمريز المتسلسل الفوري أو المتزامن). في حالة الرنا الكمي ، يكون القالب هو الدنا المكمل (cDNA) ، والذي يتم الحصول عليه عن طريق النسخ العكسي للرنا (RNA). في هذه الحالة التقنية المستخدمة هي RT-PCR الكمي أو Q-RT-PCR الكمي.
يعد تفاعل الپوليمريز المتسلسل الكمي و مصفوفة الدنا الدقيقة منهجيات حديثة لدراسة التعبير الجيني . تم استخدام الطرق الأقدم لقياس وفرة مرسال الحمض النووي الريبي: العرض التفريقي ، مقايسة حماية ريبونزكلياز واللطخة الشمالية . غالبًا ما يتم استخدام اللطخة الشمالية لتقدير مستوى التعبير عن الجين من خلال تصور وفرة نسخة مرسال الحمض النووي الريبي في عينة. في هذه الطريقة ، يتم فصل الحمض النووي الريبي المنقى عن طريق الرحلان الكهربائي للهلام أجاروز، ويتم نقله إلى مصفوفة صلبة (مثل غشاء النايلون) ، ويتم فحصه باستخدام مسبار الدنا أو الرنا معين مكمل للجين محل الاهتمام. على الرغم من أن هذه التقنية لا تزال تستخدم لتقييم التعبير الجيني ، إلا أنها تتطلب كميات كبيرة نسبيًا من الحمض النووي الريبي وتوفر فقط معلومات نوعية أو شبه كمية لمستويات مرسال الحمض الريبي النووي.[4] يمكن أن تكون أخطاء التقدير الناتجة عن الاختلافات في طريقة القياس نتيجة لسلامة الحمض النووي وكفاءة الإنزيم والعديد من العوامل الأخرى. لهذا السبب تم تطوير عدد من أنظمة التوحيد القياسي (غالبًا ما تسمى طرق التطبيع ). وقد تم تطوير بعضها لقياس التعبير الجيني الكلي ، ولكن الأكثر شيوعًا يهدف إلى تحديد الجين المحدد الذي تتم دراسته فيما يتعلق بجين آخر يسمى جين التطبيع ، والذي يتم اختياره لمستوى التعبير الثابت تقريبًا. غالبًا ما يتم اختيار هذه الجينات من الجينات التأسيسية لأن وظائفها المتعلقة بالبقاء الخلوي الأساسي تتضمن عادةً تعبيرًا جينيًا تأسيسيًا.[5][6] وهذا يمكّن الباحثين من الإبلاغ عن نسبة للتعبير عن جينات المرغوب بها مقسومة على تعبير جهاز التطبيع المحدد ، مما يتيح مقارنة الأولى دون معرفة مستوى التعبير المطلق .
أكثر جينات التطبيع شيوعًا هي تلك التي ترمز للجزيئات التالية: تيوبيولين و نازعة هيدرجين الغليسيرالدهيد 3 فوسفات و ألبيومين و سيكلوفيلين و رنا الريبوسومي .[4]
المبادئ الأساسية
يتم إجراء تفاعل الپوليمريز المتسلسل (PCR) بالزمن الحقيقي في جهاز تدوير حراري مع القدرة على إضاءة كل عينة بشعاع من الضوء بطول موجة واحد محدد على الأقل واكتشاف الفلورة المنبعثة من الفلوروفور المثار. كما أن المدور الحراري قادر أيضًا على تسخين العينات وتبريدها بسرعة ، وبالتالي الاستفادة من الخصائص الفيزيائية الكيميائية للأحماض النووية و بوليمريز الدناتتكون عملية تفاعل الپوليمريز المتسلسل (PCR) بشكل عام من سلسلة من التغيرات في درجات الحرارة التي تتكرر 25-50 مرة. تتكون هذه الدورات عادة من ثلاث مراحل: الأولى ، حوالي 95° C ، يسمح بفصل السلسلة المزدوجة للحمض النووي ؛ الثاني ، عند درجة حرارة حوالي 50-60 ° C ، يسمح بربط المشرعات بقالب دنا;[7] الثالث بين 68-72° C ، يسهل البلمرة التي يقوم بها بلمرة الدنا. نظرًا لصغر حجم الأجزاء ، عادةً ما يتم حذف الخطوة الأخيرة في هذا النوع من تفاعل الپوليمريز المتسلسل حيث يكون الإنزيم قادرًا على زيادة عددها أثناء التغيير بين مرحلة المحاذاة ومرحلة الإفساد. بالإضافة إلى ذلك ، في تفاعل الپوليمريز المتسلسل من أربع خطوات ، يتم قياس الفلورة خلال مراحل درجة الحرارة القصيرة التي تستمر فقط بضع ثوانٍ في كل دورة ، مع درجة حرارة ، على سبيل المثال ، 80° C ، لتقليل الإشارة الناتجة عن وجود ديمر المشرع عند استخدام صبغة غير محددة..[8] تعتمد درجات الحرارة والتوقيتات المستخدمة لكل دورة على مجموعة متنوعة من المعلمات ، مثل: الإنزيم المستخدم لتوليف الحمض النووي ، وتركيز الأيونات ثنائية التكافؤ و ديوكسي ريبونيوكليوتيد (dNTPs) في التفاعل ودرجة حرارة الترابط للمشرعات .[9]
التصنيف الكيميائي
يمكن تصنيف تقنية تفاعل الپوليمريز المتسلسل بالزمن الحقيقي من خلال الكيمياء المستخدمة للكشف عن منتج تفاعل الپوليمريز المتسلسل ، فلوروكرومات محددة أو غير محددة.
كشف غير محدد: تفاعل الپوليمريز المتسلسل بالزمن الحقيقي مع أصباغ مرتبطة بالحمض النووي مزدوج الشريط كمراسلين
ترتبط الصبغة الملزمة بالحمض النووي بجميع الحمض النووي مزدوج الشريط في تفاعل الپوليمريز المتسلسل ، مما يزيد من العائد الكمي للصبغة. وبالتالي فإن زيادة منتج الدنا أثناء تفاعل الپوليمريز المتسلسل (PCR) تؤدي إلى زيادة في شدة الفلورة المقاسة في كل دورة. ومع ذلك ، فإن أصباغ الدنا مزدوج الشريط مثل SYBR الخضراء ستلزم جميع نواتج تفاعل الپوليمريز المتسلسل للدنا مزدوج الشريط ، بما في ذلك نواتج تفاعل الپوليمريز المتسلسل غير النوعية (مثل مثنوي (ديمر) المشرع ). يمكن أن يتداخل هذا مع ، أو يمنع ، المراقبة الدقيقة لتسلسل الهدف المقصود.
في تفاعل الپوليمريز المتسلسل بالزمن الحقيقي مع أصباغ الدنا مزدوج الشريط يتم تحضير التفاعل كالمعتاد ، مع إضافة صبغة الدنا مزدوج الشريط الفلورية. ثم يتم تشغيل التفاعل في جهاز تفاعل الپوليمريز المتسلسل بالزمن الحقيقي ، وبعد كل دورة ، يتم قياس شدة الفلورة باستخدام كاشف ؛ تتألق الصبغة فقط عندما تكون مرتبطة ب الدنا مزدوج الشريط (أي منتج تفاعل الپوليمريز المتسلسل). تتميز هذه الطريقة بأنها تحتاج فقط إلى زوج من المشرعات لإجراء التضخيم ، مما يقلل التكاليف ؛ يمكن رصد تسلسلات هدف متعددة في أنبوب باستخدام أنواع مختلفة من الأصباغ.
كشف محدد: طريقة استخدام مسبار الفلوروسنت كمراسل
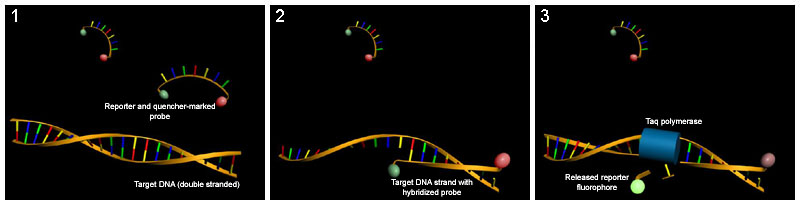
مسابير مراسل الفلورسنت تكتشف فقط الحمض النووي الذي يحتوي على تسلسل مكمل للمسبار ؛ لذلك ، فإن استخدام مسبار المراسل يزيد بشكل كبير من النوعية ، ويمكّن من تنفيذ التقنية حتى في وجود الدنا مزدوج الشريط أخرى. باستخدام اوسمة مختلفة الألوان ، يمكن استخدام مسابير الفلورسنت في فحوصات متعددة الإرسال لمراقبة عدة تسلسلات مستهدفة في نفس الأنبوب. كما أن نوعية مسابير مراسل الفلورسنت تمنع تداخل القياسات التي تسببها ديمر المشرع ، وهي نواتج ثانوية محتملة غير مرغوب فيها في تفاعل الپوليمريز المتسلسل. ومع ذلك ، تحقيقات مراسل الفلورسنت لا تمنع التأثير المثبط لديمر المشرع ، والتي قد تقلل من تراكم النواتج المرغوبة في التفاعل.
تعتمد الطريقة على مسبار قائم على الحمض النووي مع مراسل الفلورسنت في أحد طرفيه وإخماد الفلورة في الطرف المقابل من المسبار. القرب الشديد للمراسل من المخمد يمنع الكشف عن فلورته. إن تحطيم المسبار من خلال نشاط نوكلياز خارجي من 5 إلى 3 پوليمريز المستحرة المائية يكسر تقارب المراسل-المخمد وبالتالي يسمح بانبعاث فلورة، والذي يمكن الكشف عنه بعد الإثارة بالليزر. وبالتالي فإن الزيادة في المنتج المستهدف من قبل مسبار المراسل في كل دورة تفاعل الپوليمريز المتسلسل يؤدي إلى زيادة متناسبة في الفلورة بسبب انهيار المسبار وإطلاق المراسل.
- يتم تحضير تفاعل الپوليمريز المتسلسل كالمعتاد (انظر PCR ) ، ويتم إضافة مسبار المراسل.
- مع بدء التفاعل ، خلال مرحلة التلدين لتفاعل الپوليمريز المتسلسل ، يقوم المسبار والمشرعات بالتلدين إلى الحمض النووي المتسهدف.
- تبدأ البلمرة لشريط جديد من الحمض النووي من المشرعات ، وبمجرد وصول البلمرة إلى المسبار ، يحلل نوكليازها 5'-3 'المسبار ، ويفصل فيزيائيًا مراسل الفلورسنت عن المخمد ، مما يؤدي إلى زيادة في الفلورة.
- يتم الكشف عن الفلورة وقياسه في آلة تفاعل الپوليمريز المتسلسل بالزمن الحقيقي ، ويتم استخدام الزيادة الهندسية المقابلة للزيادة الأسية للمنتج لتحديد دورة القياس (C q ) في كل تفاعل.
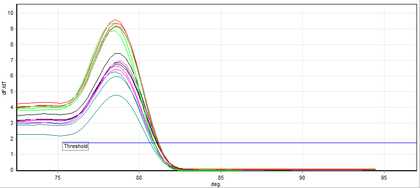
تحليل درجة حرارة الانصهار
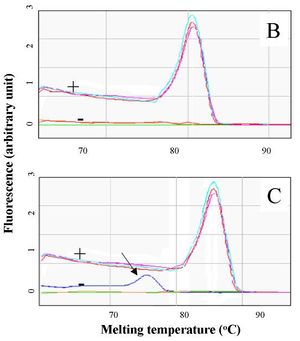
يسمح تفاعل الپوليمريز المتسلسل بالزمن الحقيقي بتحديد أجزاء محددة ومضخمة من الحمض النووي باستخدام تحليل درجة حرارة انصهارها (وتسمى أيضًا قيمة T m ، من melting temperature). الطريقة المستخدمة هي عادة تفاعل الپوليمريز المتسلسل (PCR) مع أصباغ ملزمة مزدوجة الحمض النووي كمراسلين وعادة ما تكون الصبغة المستخدمة هي SYBR الخضراء. درجة حرارة انصهار الحمض النووي خاصة بالجزء المتضخم. يتم الحصول على نتائج هذه التقنية من خلال مقارنة منحنيات التفكك لعينات الحمض النووي التي تم تحليلها.[11]
على عكس تفاعل الپوليمريز المتسلسل التقليدي ، تتجنب هذه الطريقة الاستخدام السابق لتقنيات الرحلان الكهربائي لتوضيح نتائج جميع العينات. هذا لأنه ، على الرغم من كونه تقنية حركية ، يتم عادة تقييم تفاعل الپوليمريز المتسلسل الكمي عند نقطة نهاية مميزة. وبالتالي فإن هذه التقنية عادة ما توفر نتائج أسرع و / أو تستخدم مواد تفاعلية أقل من الرحلان الكهربائي. إذا كان هناك حاجة إلى الرحلان الكهربائي اللاحق ، فمن الضروري فقط اختبار تلك العينات التي أثبتت تفاعل الپوليمريز المتسلسل بالزمن الحقيقي أنها مشكوك فيها و / أو التصديق على نتائج العينات التي ثبت أنها إيجابية لمحدِّد معين.
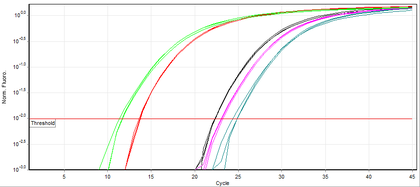
النمذجة
على عكس تفاعل الپوليمريز المتسلسل للنقطة النهائية (تفاعل الپوليمريز المتسلسل التقليدي) ، يسمح تفاعل الپوليمريز المتسلسل بالزمن الحقيقي بمراقبة المنتج المطلوب في أي وقت في عملية التضخيم عن طريق قياس الفلورة (في الإطار الزمني الحقيقي ، يتم القياس من مستواه على درجة معينة). طريقة شائعة الاستخدام لقياس كمية الحمض النووي من خلال تفاعل الپوليمريز المتسلسل بالزمن الحقيقي تعتمد على رسم فلورة مقابل عدد الدورات على مقياس لوغاريتمي . يتم تعيين درجة للكشف عن فلورة أساس الحمض النووي 3-5 مرات الانحراف المعياري لضجيج الإشارة فوق الخلفية. يسمى عدد الدورات التي يتجاوز فيها الفلورة الدرجة دورة الدرجة (C t ) أو ، وفقًا لإرشادات MIQE ، دورة القياس (C q ).[12]
خلال مرحلة التضخيم الأسي ، تضاعف كمية قالب الحمض النووي المستهدف (الأمبليكون) كل دورة. على سبيل المثال ، تحتوي عينة الحمض النووي التي تسبق C q عينة أخرى بثلاث دورات على 2 3 = 8 أضعاف القالب. ومع ذلك ، غالبًا ما تكون كفاءة التضخيم متغيرة بين المشرعات والقوالب. لذلك ، يتم تقييم كفاءة تركيبة قالب المشرع في تجربة المعايرة بالتخفيفات التسلسلية لنموذج الدنا لإنشاء منحنى قياسي للتغيير في (C q ) مع كل تخفيف. ثم يتم استخدام منحدر الانكفاء الخطي لتحديد كفاءة التضخيم ، والتي تكون 100 ٪ إذا أدى التخفيف بنسبة 1: 2 إلى اختلاف (C q ) قدره 1. تقدم طريقة درجة الدورة العديد من الافتراضات لآلية التفاعل ولها اعتماد على البيانات من مناطق منخفضة الإشارة إلى الضوضاء لملف التضخيم الذي يمكن أن يقدم تباينًا كبيرًا أثناء تحليل البيانات.[13]
لقياس التعبير الجيني ، يتم طرح (C q ) للرنا أو الدنا من الجين محل الاهتمام من (C q ) للرنا / الدنا من الجين التأسيسي في نفس العينة للتطبيع للاختلاف في كمية ونوعية الرنا بين عينات مختلفة. يُطلق على إجراء التطبيع هذا طريقة ΔCt-method [14]ويسمح بمقارنة التعبير عن الجين محل الاهتمام بين العينات المختلفة. ومع ذلك ، لمثل هذه المقارنة ، يجب أن يكون التعبير عن الجين المرجعي التطبيع متشابهًا جدًا عبر جميع العينات. وبالتالي ، فإن اختيار جين مرجعي يفي بهذا المعيار له أهمية كبيرة ، وغالبًا ما يمثل تحديًا ، لأن عددًا قليلاً جدًا من الجينات يظهر مستويات متساوية من التعبير عبر مجموعة من الظروف أو الأنسجة المختلفة. .[15][16] لى الرغم من أن تحليل درجة الدورة يتكامل مع العديد من أنظمة البرامج التجارية ، إلا أن هناك طرقًا أكثر دقة وموثوقية لتحليل بيانات ملف تضخيم التي يجب أخذها في الاعتبار في الحالات التي تكون فيها إمكانية التكرار مصدر قلق .[13]
كما تم اقتراح طرق تحديد كمية qPCR القائمة على الآلية ، ولها ميزة أنها لا تتطلب منحنى قياسي للقياس الكمي. وقد ثبت أن طرقًا مثل MAK2 [17] تتمتع بأداء كمي مساوٍ أو أفضل لطرق المنحنى القياسي. تستخدم هذه الأساليب القائمة على الآلية المعرفة حول عملية تضخيم البلمرة لتوليد تقديرات لتركيز العينة الأصلي. يتضمن امتداد هذا النهج نموذجًا دقيقًا لملف تعريف تفاعل الپوليمريز المتسلسل بالكامل ، والذي يسمح باستخدام بيانات عالية الإشارة إلى الضوضاء والقدرة على التحقق من جودة البيانات قبل التحليل .[13]
وفقا لبحث رويجتر وآخرون. .[18] يفترض MAK2 كفاءة تضخيم ثابتة أثناء تفاعل الپوليمريز المتسلسل. ومع ذلك ، فقد أظهر التحليل النظري لتفاعل الپوليمريز المتسلسل ، الذي تم اشتقاق منه MAK2 ، أن كفاءة التضخيم ليست ثابتة طوال تفاعل الپوليمريز المتسلسل. بينما يوفر قياس MAK2 تقديرات موثوقة لتركيز الحمض النووي المستهدف في عينة في ظل ظروف qPCR العادية ، فإن MAK2 لا يعول بشكل موثوق على تركيز الهدف لفحوصات qPCR مع المنافسين .
التطبيقات
هناك العديد من التطبيقات لتفاعل الپوليمريز المتسلسل الكمي في المختبر . يتم استخدامه عادة لكل من البحث التشخيصي والأساسي . تشمل استخدامات التقنية في الصناعة تحديد كمية الحمل الميكروبي في الأطعمة أو في المواد النباتية ، والكشف عن GMOs ( الكائنات المعدلة وراثيًا ) وتحديد الأنواع الجينية لمسببات الأمراض الفيروسية البشرية وتنميتها.
القياس الكمي للتعبير الجيني
لا يمكن الاعتماد على القياس الجيني للتعبير عن طريق طرق الكشف التقليدية للحمض النووي. اكتشاف مرسال الحمض النووي الريبي على لطخة شمالية أو نواتج تفاعل الپوليمريز المتسلسل على هلام أو لطخة جنوبية لا يسمح بتحديد دقيق. .[19] على سبيل المثال ، على مدى 20-40 دورة من تفاعل الپوليمريز المتسلسل نموذجي ، تصل كمية منتج الدنا إلى هضبة لا ترتبط مباشرة بكمية الدنا المستهدفة في تفاعل الپوليمريز المتسلسل الأولي .[20]
يمكن استخدام تفاعل الپوليمريز المتسلسل بالزمن الحقيقي لقياس الأحماض النووية من خلال طريقتين شائعتين: القياس الكمي النسبي والكمي المطلق .[21] يعطي التحديد الكمي المطلق العدد الدقيق لجزيئات الحمض النووي المستهدفة بالمقارنة مع معايير الحمض النووي باستخدام منحنى المعايرة . لذلك من الضروري أن يكون تفاعل الپوليمريز المتسلسل للعينة والمعيار لهما نفس كفاءة التضخيم.[22] يعتمد القياس الكمي على الجينات المرجعية الداخلية لتحديد اختلافات الطية في التعبير عن الجين المستهدف. يتم التعبير عن القياس الكمي على أنه التغير في مستويات التعبير عن مرسال الحمض النووي الريبي يفسر على أنه دنا مكمل (cDNA ، ولدت عن طريق النسخ العكسي لمرسال الحمض النووي الريبي). من السهل إجراء القياس الكمي لأنه لا يتطلب منحنى معايرة حيث يتم مقارنة كمية الجين المدروس مع كمية الجين المرجعي للتحكم
بما أن الوحدات المستخدمة للتعبير عن نتائج القياس الكمي غير مهمة ، يمكن مقارنة النتائج عبر عدد من RTqPCR المختلفة. السبب وراء استخدام واحد أو أكثر من جينات التأسيسة هو تصحيح الاختلاف الغير محدد ، مثل الاختلافات في كمية ونوعية الحمض النووي الريبي المستخدم ، والتي يمكن أن تؤثر على كفاءة النسخ العكسي وبالتالي عملية تفاعل الپوليمريز المتسلسل بأكملها. ومع ذلك ، فإن الجانب الأكثر أهمية في العملية هو أن الجين المرجعي يجب أن يكون مستقرًا .[23]
تم اختيار هذه الجينات المرجعية تقليديا في علم الأحياء الجزيئي باستخدام دراسات نوعية أو شبه كمية مثل الفحص البصري لهلام الرنا أو قياس كثافة اللطخة الشمالية أو تفاعل الپوليمريز المتسلسل شبه الكمي (محاكاة PCR). الآن ، في عصر الجينوم ، من الممكن إجراء تقدير أكثر تفصيلاً للعديد من الكائنات الحية باستخدام تقنيات النسخ.[24] ومع ذلك ، فقد أظهرت الأبحاث أن تضخيم غالبية الجينات المرجعية المستخدمة في تحديد التعبير عن مرسال الحمض النووي الريبي يختلف باختلاف الظروف التجريبية .[25][26][27] لذلك من الضروري إجراء دراسة منهجية أولية سليمة إحصائيا من أجل اختيار الجين المرجعي الأكثر ملاءمة .
تم تطوير عدد من الخوارزميات الإحصائية التي يمكنها اكتشاف الجين أو الجينات الأكثر ملاءمة للاستخدام في ظل ظروف معينة. يمكن لمثل geNORM أو BestKeeper مقارنة الأزواج أو المتوسط الهندسي لمصفوفة من الجينات والأنسجة المرجعية المختلفة. [28][29]
الاستخدامات التشخيصية
يتم تطبيق تفاعل الپوليمريز المتسلسل التشخيصي النوعي للكشف السريع عن الأحماض النووية التي يتم تشخيصها ، على سبيل المثال ، الأمراض المعدية و السرطان والتشوهات الجينية. أدى إدخال اختبارات تفاعل الپوليمريز المتسلسل النوعية إلى مختبر علم الأحياء الدقيقة السريري إلى تحسن كبير في تشخيص الأمراض المعدية,[30] ويتم نشرها كأداة للكشف عن الأمراض الناشئة حديثًا ، مثل سلالات جديدة من الإنفلونزا و ڤيروس كورونا ، في الاختبارات التشخيصية .[31][32]
الاستخدامات الميكروبيولوجية
يستخدم تفاعل الپوليمريز المتسلسل الكمي أيضًا من قبل علماء الأحياء الدقيقة الذين يعملون في مجالات سلامة الأغذية وتلف الأغذية والتخمير وتقييم المخاطر الميكروبية لجودة المياه (مياه الشرب والترفيه) وفي حماية الصحة العامة .[33]
يمكن أيضًا استخدام qPCR لتضخيم العلامات التصنيفية أو الوظيفية للجينات في الحمض النووي المأخوذة من العينات البيئية .[34] يتم تمثيل العلامات بواسطة أجزاء جينية من الحمض النووي أو الحمض النووي التكميلي .[34] من خلال تضخيم عنصر جيني معين ، يمكن للمرء تحديد كمية العنصر في العينة قبل التضخيم. .[34]يمكن أن يساعد استخدام العلامات التصنيفية (الجينات الريبوزومية) و qPCR في تحديد كمية الكائنات الحية الدقيقة في العينة ، ويمكن تحديد العائلات أو الأجناس أو الأنواع المختلفة بناءً على نوعية العلامة .[34] يمكن أن يظهر استخدام العلامات الوظيفية (جينات ترميز البروتين) تعبيرًا جينيًا داخل المجتمع ، مما قد يكشف عن معلومات حول البيئة .[34]
الكشف عن مسببات المرض (الممراض)
تسعى الصناعة الزراعية باستمرار إلى إنتاج غرسات نباتية أو شتلات خالية من مسببات الأمراض من أجل منع الخسائر الاقتصادية وحماية الصحة. تم تطوير أنظمة تسمح بالكشف عن كميات صغيرة من الحمض النووي ل Phytophthora ramorum ، وهو فطر بيضي يقتل سنديان والأنواع الأخرى ، ممزوجًا بالحمض النووي للنبات العائل ويستند التمييز بين الحمض النووي للممرض والنبات على تضخيم تسلسلات ITS ، والفواصل الموجودة في منطقة تشفير جين الريبوسومي للرنا ، والتي تعتبر مميزة لكل صنف.[35] كما تم تطوير إصدارات ميدانية من هذه التقنية لتحديد نفس الممراض.[36]
الكشف عن الكائنات المعدلة وراثياً
يمكن استخدام qPCR باستخدام النسخ العكسي (RT-qPCR) للكشف عن الكائنات المعدلة وراثيًا نظرًا لحساسيتها ونطاقها الدينامكي في الكشف عن الحمض النووي. عادة ما تكون البدائل مثل تحليل الحمض النووي أو البروتين أقل حساسية. يتم استخدام المشرعات المحددة التي لا تضخم الجينات المحورة ولكن المحفز أو الفاصل أو حتى التسلسلات الوسيطة المستخدمة أثناء عملية هندسة الناقل. نظرًا لأن عملية إنشاء مصنع معدّل وراثيًا تؤدي عادةً إلى إدخال أكثر من نسخة واحدة من المتحوّل الجيني ، يتم أيضًا تقييم كميته بشكل شائع. غالبًا ما يتم ذلك عن طريق القياس الكمي باستخدام جين تحكم من الأنواع المعالجة الموجودة فقط كنسخة واحدة .[37][38]
القياس السريري والتنميط الجيني
يمكن أن تكون الفيروسات موجودة في البشر بسبب العدوى المباشرة أو العدوى المشتركة مما يجعل التشخيص صعبًا باستخدام التقنيات الكلاسيكية ويمكن أن يؤدي إلى مآل وعلاج غير صحيحين. يتيح استخدام qPCR كلاً من التحديد الكمي والتنميط الجيني (توصيف السلالة ، التي تتم باستخدام منحنيات انصهار) لفيروس مثل ڤيروس التهاب الكبد الڤيروسي ب.[39] T[40]درجة العدوى ، التي يتم قياسها كنسخ الجينوم الفيروسي لكل وحدة من أنسجة المريض ، ذات صلة في كثير من الحالات ؛ على سبيل المثال ، يرتبط احتمال تنشيط فيروس الهربس البسيط من النوع الأول بعدد الخلايا العصبية المصابة في العقدة.[41] يتم إجراء هذا التحديد الكمي إما عن طريق النسخ العكسي أو بدونه ، كما يحدث إذا أصبح الفيروس مدمجًا في الجينوم البشري في أي مرحلة من دورته ، كما يحدث في حالة فيروس الورم الحليمي البشري ( HPV ) ، حيث توجد بعض متغيراته المرتبطة بظهور سرطان عنق الرحم.[42]
المراجع
- ^ Bustin SA, Benes V, Garson JA, Hellemans J, Huggett J, Kubista M, Mueller R, Nolan T, Pfaffl MW, Shipley GL, Vandesompele J, Wittwer CT (2009). "The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments". Clinical Chemistry. 55 (4): 611–622. doi:10.1373/clinchem.2008.112797. PMID 19246619.
- ^ Logan, Julie; Edwards, Kirstin; Saunders, Nick, eds. (2009). Real-Time PCR: Current Technology and Applications. Caister Academic Press. ISBN 978-1-904455-39-4.
{{cite book}}: Unknown parameter|last-author-amp=ignored (|name-list-style=suggested) (help) - ^ Watson, J D; Baker, T A; Bell, S P; Gann, A; Levine, M; Losick, R (2004). Molecular Biology of the Gene (Fifth ed.). San Francisco: Benjamin Cummings. ISBN 978-0-321-22368-5.
- ^ أ ب Michael W. Pfaff, Ales Tichopad, Christian Prgomet and Tanja P. Neuvians (2005). Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper – Excel-based tool using pair-wise correlations Biotechnology Letters 26:509–515
- ^ Pfaffl, MW; Horgan, GW; Dempfle, L (2002). "Relative Expression Software Tool (REST©) for group wise comparison and statistical analysis of relative expression results in real-time PCR". Nucleic Acids Res. 30 (9): e36. doi:10.1093/nar/30.9.e36. PMC 113859. PMID 11972351.
- ^ Vandesompele, J; De Preter, K; Pattyn, F; Poppe, B; Van Roy, N; De Paepe, A; Speleman, F (2002). "Accurate normalisation of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes". Genome Biology. 3 (7): 1–12. doi:10.1186/gb-2002-3-7-research0034. PMC 126239. PMID 12184808.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Rychlik W, Spencer WJ, Rhoads RE (1990). "Optimization of the annealing temperature for DNA amplification in vitro". Nucleic Acids Res. 18 (21): 6409–6412. doi:10.1093/nar/18.21.6409. PMC 332522. PMID 2243783.
- ^ Pfaffl, Michael (2000). "Development and Validation of an Externally Standardised Quantitative Insulin-like Growth Factor-1 RT-PCR Using LightCycler SYBR Green I Technology" (PDF). Biochemica (3) – via gene-quantification.org.
- ^ Joseph Sambrook; David W. Russel (2001). Molecular Cloning: A Laboratory Manual (3rd ed.). Cold Spring Harbor, N.Y.: Cold Spring Harbor Laboratory Press. ISBN 978-0-87969-576-7.
{{cite book}}: Unknown parameter|lastauthoramp=ignored (|name-list-style=suggested) (help) - ^ Ponchel F; Toomes C; Bransfield K; Leong F.T; Douglas S.H; Field S.L; Bell S.M; Combaret V; Puisieux A; Mighell A.J (2003). "Real-time PCR based on SYBR-Green I fluorescence: An alternative to the TaqMan assay for a relative quantification of gene rearrangements, gene amplifications and micro gene deletions". BMC Biotechnol. 3: 18. doi:10.1186/1472-6750-3-18. PMC 270040. PMID 14552656.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Ririe K.M; Rasmussen R.P; Wittwer C.T. (1997). "Product Differentiation by Analysis of DNA Melting Curves during the Polymerase Chain Reaction" (PDF). Analytical Biochemistry. 245 (2): 154–160. doi:10.1006/abio.1996.9916. PMID 9056205.
- ^ Stephen A. Bustin; Vladimir Benes; Jeremy A. Garson; Jan Hellemans; Jim Huggett; Mikael Kubista; Reinhold Mueller; Tania Nolan; Michael W. Pfaffl; Gregory L. Shipley; Jo Vandesompele; Carl T. Wittwer (April 2009). "The MIQE Guidelines: Minimum Information for Publication of Quantitative Real-Time PCR Experiments". Clin. Chem. 55 (4): 611–622. doi:10.1373/clinchem.2008.112797. PMID 19246619.
{{cite journal}}: Unknown parameter|last-author-amp=ignored (|name-list-style=suggested) (help) - ^ أ ب ت Carr, A. C.; Moore, S. D. (2012). Lucia, Alejandro (ed.). "Robust Quantification of Polymerase Chain Reactions Using Global Fitting". PLOS One. 7 (5): e37640. Bibcode:2012PLoSO...737640C. doi:10.1371/journal.pone.0037640. PMC 3365123. PMID 22701526.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Schefe JH, Lehmann KE, Buschmann IR, Unger T, Funke-Kaiser H (2006). "Quantitative real-time RT-PCR data analysis: current concepts and the novel "gene expression's CT difference" formula". J Mol Med. 84 (11): 901–910. doi:10.1007/s00109-006-0097-6. PMID 16972087.
- ^ Nailis H, Coenye T, Van Nieuwerburgh F, Deforce D, Nelis HJ (2006). "Development and evaluation of different normalization strategies for gene expression studies in Candida albicans biofilms by real-time PCR". BMC Mol. Biol. 7 (1): 25. doi:10.1186/1471-2199-7-25. PMC 1557526. PMID 16889665.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Nolan T, Hands RE, Bustin SA (2006). "Quantification of mRNA using real-time RT-PCR". Nat. Protoc. 1 (3): 1559–1582. doi:10.1038/nprot.2006.236. PMID 17406449.
- ^ Boggy G, Woolf PJ (2010). Ravasi T (ed.). "A Mechanistic Model of PCR for Accurate Quantification of Quantitative PCR Data". PLOS One. 5 (8): e12355. Bibcode:2010PLoSO...512355B. doi:10.1371/journal.pone.0012355. PMC 2930010. PMID 20814578.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Ruijter JM, Pfaffl MW, Zhao S, Spiess AN, Boggy G, Blom J, Rutledge RG, Sisti D, Lievens A, De Preter K, Derveaux S, Hellemans J, Vandesompele J (2012). "Evaluation of qPCR curve analysis methods for reliable biomarker discovery: bias, resolution, precision, and implications". Methods. 59 (1): 32–46. doi:10.1016/j.ymeth.2012.08.011. PMID 22975077.
- ^ Bruce Gelerter. "PEMF For Treatment Of Corneal Disorders". lemuriatechnologies.com. Archived from the original on 2014-06-09.
- ^ Overbergh, L.; Giulietti, A.; Valckx, D.; Decallonne, R.; Bouillon, R.; Mathieu, C. (2003). "The use of real-time reverse transcriptase PCR for the quantification of cytokine gene expression". Journal of Biomolecular Techniques : JBT. 14 (1): 33–43. PMC 2279895. PMID 12901609.
- ^ S. Dhanasekaran; T. Mark Doherty; John Kenneth; TB Trials Study Group (March 2010). "Comparison of different standards for real-time PCR-based absolute quantification". Journal of Immunological Methods. 354 (1–2): 34–39. doi:10.1016/j.jim.2010.01.004. PMID 20109462.
- ^ Bar, Tzachi; Kubista, Mikael; Tichopad, Ales (2011-10-19). "Validation of kinetics similarity in qPCR". Nucleic Acids Research (in الإنجليزية). 40 (4): 1395–1406. doi:10.1093/nar/gkr778. ISSN 0305-1048. PMC 3287174. PMID 22013160.
- ^ Brunner, AM; Yakovlev, IA; Strauss, SH (2004). "Validating internal controls for quantitative plant gene expression studies". BMC Plant Biol. 4: 14. doi:10.1186/1471-2229-4-14. PMC 515301. PMID 15317655. Archived from the original on 2013-08-02.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ McGettigan, Paul A (2013). "Transcriptomics in the RNA-seq era". Current Opinion in Chemical Biology. 17 (1): 4–11. doi:10.1016/j.cbpa.2012.12.008. PMID 23290152.
- ^ Thellin, O; Zorzi, W; Lakaye, B; De Borman, B; Coumans, B; Henne, G; Grisar, T; Igout, A; Heinen, E (1999). "Housekeeping genes as internal standards: use and limits". J Biotechnol. 75 (2–3): 197–200. doi:10.1016/s0168-1656(99)00163-7. PMID 10617337.
- ^ Radonic, A; Thulke, S; Mackay, IM; Landt, O; Siegert, W; Nitsche, A (2004). "Guideline for reference gene selection for quantitative real-time PCR". Biochem Biophys Res Commun. 313 (4): 856–862. doi:10.1016/j.bbrc.2003.11.177. PMID 14706621. Archived from the original on 2013-08-02.
- ^ Dheda, K; Huggett, JF; Bustin, SA; Johnson, MA; Rook, G; Zumla, A (2004). "Validation of housekeeping genes for normalizing RNA expression in real-time PCR". BioTechniques. 37 (1): 112–119. doi:10.2144/04371RR03. PMID 15283208. Archived from the original on 2013-08-02.
- ^ Vandesompele J, De Preter K, Pattyn F, Poppe B, Van Roy N, De Paepe A, Speleman F (2002) Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes" Genome Biol 37: RESEARCH0034
- ^ Pfaffl, MW; Tichopad, A; Prgomet, C; Neuvians, TP (2004). "Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper—Excel-based tool using pair-wise correlations". Biotechnol Lett. 26 (6): 509–515. doi:10.1023/b:bile.0000019559.84305.47. PMID 15127793. Archived from the original on 2013-08-02.
- ^ Espy, M.J. (January 2006). "Real-Time PCR in Clinical Microbiology: Applications for Routine Laboratory Testing". Clinical Microbiology Reviews. 19 (3): 165–256. doi:10.1128/CMR.19.1.165-256.2006. PMC 1360278. PMID 16418529.
- ^ "FDA-cleared RT-PCR Assays and Other Molecular Assays for Influenza Viruses" (PDF). cdc.gov.
- ^ "rRT-PCR, a method to confirm Wuhan coronavirus case – Artificial Intelligence for Chemistry" (in الإنجليزية الأمريكية). Retrieved 2020-01-26.
- ^ Filion, M, ed. (2012). Quantitative Real-time PCR in Applied Microbiology. Caister Academic Press. ISBN 978-1-908230-01-0.
- ^ أ ب ت ث ج Bouchez, Blieux, Dequiedt, Domaizon, Dufresne, Ferreira, Godon, Hellal, Joulian, Quaiser, Martin-Laurent, Mauffret, Monier, Peyret, Schmitt-Koplin, Sibourg, D’oiron, Bispo, Deportes, Grand, Cuny, Maron, Ranjard (September 2016). "Molecular Microbiology Methods For Environmental Diagnosis". Environmental Chemistry Letters. 14 (4): 423-441. doi:10.1007/s10311-016-0581-3. Retrieved 11 May 2020.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ^ Baldwin, B.G. (1992). "Phylogenetic utility of the internal transcribed spacers of nuclear ribosomal DNA in plants: An example from the Compositaogy". Molecular Phylogenetics and Evolution. 1 (1): 3–16. doi:10.1016/1055-7903(92)90030-K. PMID 1342921.
- ^ Tomlinson, J. A.; Barker, I.; Boonham, N. (2007). "Faster, Simpler, More-Specific Methods for Improved Molecular Detection of Phytophthora ramorum in the Field". Applied and Environmental Microbiology. 73 (12): 4040–4047. doi:10.1128/AEM.00161-07. PMC 1932743. PMID 17449689.
- ^ Holst-Jensen, Arne; Rønning, Sissel B.; Løvseth, Astrid; Berdal, Knut G. (2003). "PCR technology for screening and quantification of genetically modified organisms (GMOs)". Analytical and Bioanalytical Chemistry. 375 (8): 985–993. doi:10.1007/s00216-003-1767-7. PMID 12733008.
- ^ Brodmann P.D; Ilg E.C; Berthoud H; Herrmann A. (2002). "… -Time Quantitative Polymerase Chain Reaction Methods for Four Genetically Modified Maize Varieties …". Journal of AOAC International. 85 (3): 646–653. doi:10.1093/jaoac/85.3.646. PMID 12083257.
- ^ Yeh S.H. Tsai C.Y. Kao J.H. Liu C.J. Kuo T.J. Lin M.W. Huang W.L. Lu S.F. Jih J. Chen D.S. Others (2004). "Quantification and genotyping of hepatitis B virus in a single reaction by real-time PCR and melting …". Journal of Hepatology. 41 (4): 659–666. doi:10.1016/j.jhep.2004.06.031. PMID 15464248.
- ^ Yeh S.H. Tsai C.Y. Kao J.H. Liu C.J. Kuo T.J. Lin M.W. Huang W.L. Lu S.F. Jih J. Chen D.S. Others (2004). "Quantification and genotyping of hepatitis B virus in a single reaction by real-time PCR and melting …". Journal of Hepatology. 41 (4): 659–666. doi:10.1016/j.jhep.2004.06.031. PMID 15464248.
- ^ Sawtell N.M. (1998). "The Probability of in Vivo Reactivation of Herpes Simplex Virus Type 1 Increases with the Number of Latently Infected Neurons in the Ganglia". Journal of Virology. 72 (8): 6888–6892. doi:10.1128/JVI.72.8.6888-6892.1998. PMC 109900. PMID 9658140.
- ^ Peter M. Rosty C. Couturier J. Radvanyi F. Teshima H. Sastre-garau X. (2006). "MYC activation associated with the integration of HPV DNA at the MYC locus in genital tumours". Oncogene. 25 (44): 5985–5993. doi:10.1038/sj.onc.1209625. PMID 16682952.
قائمة المراجع
- Elyse; Houde, Alain (2002). "La PCR en temps réel: principes et applications" (PDF). Reviews in Biology and Biotechnology. 2 (2): 2–11. Archived from the original (PDF) on 2009-06-12.
- Bustin, SA (2000). "Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays". J Mol Endocrinol. 25 (2): 169–193. doi:10.1677/jme.0.0250169. PMID 11013345.
- Higuchi, R.; Dollinger, G.; Walsh, P.S.; Griffith, R. (1992). "Simultaneous amplification and detection of specific DNA-sequences". Bio-Technology. 10 (4): 413–417. doi:10.1038/nbt0492-413. PMID 1368485.
- Holland, P.M.; Abramson, R.D.; Watson, R.; Gelfand, D.H. (1991). "Detection of specific polymerase chain reaction product by utilizing the 50 !30 exonuclease activity of Thermus aquaticus DNA polymerase". Proc. Natl. Acad. Sci. USA. 88 (16): 7276–7280. Bibcode:1991PNAS...88.7276H. doi:10.1073/pnas.88.16.7276. JSTOR 2357665. PMC 52277. PMID 1871133.
- Kubista, M; Andrade, JM; Bengtsson, M; Forootan, A; Jonak, J; Lind, K; Sindelka, R; Sjoback, R; Sjogreen, B; Strombom, L; Stahlberg, A; Zoric, N (2006). "The real-time polymerase chain reaction". Mol. Aspects Med. 27 (2–3): 95–125. doi:10.1016/j.mam.2005.12.007. PMID 16460794.
- Higuchi, R.; Fockler, C.; Dollinger, G.; Watson, R. (1993). "Kinetic PCR: Real time monitoring of DNA amplification reactions". Biotechnology. 11 (9): 1026–1030. doi:10.1038/nbt0993-1026. PMID 7764001.
- Filion, M. (2012). Quantitative Real-time PCR in Applied Microbiology. Caister Academic Press. ISBN 978-1-908230-01-0.
- Wawrik, B; Paul, JH; Tabita, FR (2002). "Real-time PCR quantification of rbcL (ribulose-1,5-bisphosphate carboxylase/oxygenase) mRNA in diatoms and pelagophytes". Appl. Environ. Microbiol. 68 (8): 3771–3779. doi:10.1128/aem.68.8.3771-3779.2002. PMC 123995. PMID 12147471.
- Logan J; Edwards K; Saunders N, eds. (2009). Real-Time PCR: Current Technology and Applications. Caister Academic Press. ISBN 978-1-904455-39-4.