عرض الآكلة
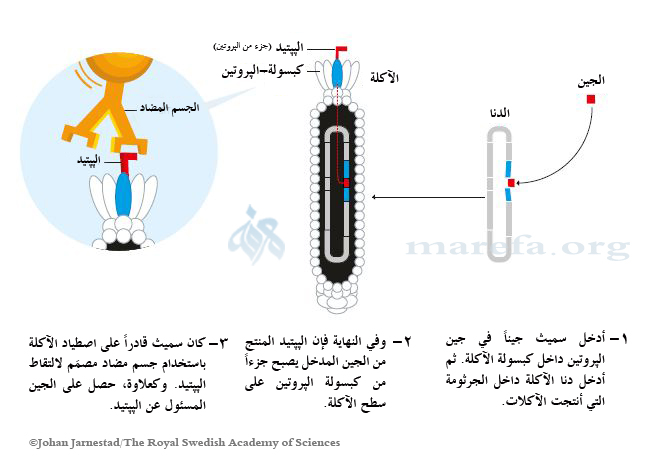
عرض الآكلة أو عرض العاثية Phage display، هي تقنية معملية لدراسة تفاعلات الپروتين-پروتين، الپروتين-الپپتيد، والپروتين-الدنا التي تستخدم الآكلات (الڤيروسات التي تصيب الجراثيم) لتوصيل الپروتينات بالمعلومات الوراثية التي ترمزها.[1] في هذه التقنية، يتم غرس الجين ترميز الپروتين المعني داخل جين الپروتين المغلف للآكلات، مما يؤدي إلى عرض الپروتين في الخارج بينما يتم احتواء جين الپروتين في الداخل، مما يؤدي إلى الاتصال بين النمط الجيني والنمط الظاهري. بعد ذلك يمك فحص هذه الآكلات العارضة بحثاً عن پروتينات، پپتيدات أو تسلسلات أحماض أمينية أخرى، من أجل التحقق من التفاعل بين الپروتين المعروض وهذه الجزيئات الأخرى. على هذا النحو، يمكن فحص مكتبات الپروتينات وتضخيمها في عملية تسمى الانتخاب المعملي، مناظرة للانتخاب الطبيعي.
ومن أكثر الآكلات شيوعاً في عرض الآكلة M13 والآكلة الفتيلية fd ،[2][3] على الرغم من أن الآكلة T4،[4] T7، و λ يتم استخدامها أيضاً.
التاريخ
وُصف عرض الآكلة لأول مرة بواسطة جورج سميث عام 1985، عندما أظهر الپپتيدات على آكلة فتيلية Filamentous phage بدمج الپپتيد[مطلوب توضيح] المعني داخل الجين الثالث لآكلة فتيلية.[1] ويزعم أن جورج پچنك يملك براءة اختراع قبل عام 1985 تصف أيضاً توليد مكتبات من عروض الآكلة.[5] خضعت هذه التقنية للمزيد من التطوير والتحسين بواسطة مجموعات بحثية في معمل الأحياء الجزيئية مع گرگ ونتر وجورج مكافرتي، معهد سكريپس للأبحاث مع لرنر وبارباس والمركز الألماني لأبحاث السرطان مع برايتلنگ ودوبل لعرض پروتينات مثل الأجسام المضادة لهندسة الپروتين العلاجية. حاز سميث وونتر مناصفة جائزة نوبل في الكيمياء 2018 عن إسهامهما في تطوير عرض الآكلة.[6]
المبدأ
كما في نظام ثنائي الهجائن two-hybrid system، يُستخدم عرض الآكلة للفحص غزير المعلومات لتفاعلات الپروتين. في حالة عرض العاثية M13، فإن ترميز دنا الپروتين أو الپپتيد المعني يكون مرتبط بالجين pIII أو pVIII، ومرمز للپروتين المغلـِّف Protein of the viral capsid، على التوالي. مواقع الاستنساخ المتعددة تستخدم أحياناً لضمان دخول القطع في إطارات القراءة الثلاثة الممكنة بالكامل لذلك تكون قطعة الـcDNA مترجمة في الإطار الصحيح. بعد ذلك يتم إدخال جين الآكلة وهجين الدنا (في عملية تسمى "تتبع المسار") داخل الخلايا الجرثومية للإشريشيا المعوية (E. coli)، مثل TG1، SS320، ER2738، أو XL1-Blue. إذا كان الناقل "Phagemid" (ناقل إنشاء العرض المبسط) يستخدم جزيئات آكلة عندئذ لن ينطلق من خلايا الإشرشيا المعوية حتى تصاب بالآكلة المساعدة، التي تمكنها من تعبئة دنا الآكلة وتجميع الڤيرونات مع قطعة الپروتين المتعلقة كجزء من غلافها الخارجي سواء على الپروتين المغلف الأصغر (pIII) أو الأكبر (pVIII).
بتثبيت أهداف الدنا أو الپروتين المتعلقة على سطح صفيحة المعايرة الدقيقة بشكل جيد، ستظل الآكلة التي تعرض الپروتين المرتبط بأحد هذه الأهداف على سطحه بينما يتم إزالة الأهداف الأخرى بالغسيل. هذه الأهداف الباقية التي قد تكون مشطوفة، تستخدم لإنتاج المزيد من العاثيات (عن طريق العدوى الجرثومية بآكلة مساعدة) ولإنتاج خليط الآكلات التي يتم إثرائها بعاثيات متعلقة (أي ربطها). يشار إلى الدورة المتكررة لهذه الخطوات "بالفصل"، في إشارة إلى إثراء عينة الذهب بإزالة المواد الغير مرغوب فيها.
العاثية المشطوفة في الخطوة الأخيرة يمكن استخدامها لنقل العدوى للمضيف الجرثومي المناسب، والتي يمكن منها جمع الآكلات وتسلسلات الدنا المتعلقة المستخلصة والمتسلسلة لتحديد الپروتينات أو قطع الپروتين المتفاعلة ذات العلاقة.
التطبيقات
تتضمن تطبيقات تقنية عرض الآكلة تحديد الأطراف المتفاعلة في الپروتين (والتي يمكن استخدامها كآكلة ثابتة "طُعم" حيث تتألف مكتبة الدنا من جميع تسلسلات الترميز للخلية، النسيج أو العضية) وبالتالي يمكن تحديد وظيفة أو آلية وظيفة هذا الپروتين.[7] كما تستخدم عروض الآكلات بشكل موسع لتطوير الپروتين في المعمل (المعروف بهندسة الپروتين). وعلى هذا النحو، فإن عرض الآكلة يصبح أداة مفيدة في اكتشاف الأدوية. ويستخدم عرض الآكلة لإيجاد ربائط جديدة (مستقبلات الانزيمات، ناهضات وغرماء المستقبل) لاستهداف الپروتينات.[8][9][10] كما تستخدم هذه التقنية لتحديد مستضدات الأورام (لاستخدامها في التشخيص والتوجيه العلاجي)[11] وفي البحث عن تفاعلات الپروتين-الدنا[12] باستخدام مكتبات الدنا التي أنشئت خصيصاً لهذا الغرض بشرائح عشوائية. مؤخراً، يستخدم عرض الآكلة أيضاً في سياق علاجات السرطات- مثل نقل الخلايا للتبني.[13] في هذه الحالات، يستخدم عرض الآكلة لخلق واختيار الأجسام المضادة المخلقة التي تستهدف الپروتنات الموجودة على سطح الورم.[13] يُجرى هذا داخل المستقبلات المخلقة لخلايا تائية تم جمعها من المريض والتي تستخدم للقضاء على المرض.[14]
ومن الطرق المنافسة لتطور الپروتين في المعمل عرض الخميرة، العرض الجرثومي، عرض الريبوسوم، وعرض مرسال الرنا.
إنضاج الأجسام المضادة معملياً
أحدث عرض آكلات الأجسام المضادة ثورة في اكتشاف أدوية الأجسام المضادة. قام بالمراحل الأولى من العمل معامل في معمل علم الأحياء الجزيئي (گرگ وينتر وجون مككافرتي) ومعهد سكريپس للأبحاث (ريتشارد لرنر وكارلوس ف. بارباس) والمركز الألماني لأبحاث السرطان (فرنك برايتلينگ وستفان دوبل).[15][16][17] وفي 1991، أفادت مجموعة سكريپسأول عرض (لآكلة) واختيار الأجسام المضادة على تلك الآكلة.[18] وصفت هذه الدراسة الأولية العزل السريع (لشظية ربط المستضد Fragment antigen-binding - FAB التي ربطت ذيفان التيتانوس tetanus toxin وسرعان ما تم مد الطريقة لتُستَخدم لإستنتساخ للأجسام المضادة البشرية anti-HIV-1 لتصميم لقاح وعلاج المرض.[19][20][21][22][23]
أصبحت مكتبات عرض الآكلة للأجسام المضادة طريقة فعالة لدراسة الاستجابة المناعية وأيضاً كوسيلة للاختيار السريع ولتطوير الأجسام المضادة المؤنسنة من أجل العلاج. لاحقاً أُستخدم عرض آكلة الأجسام المضادة من قبل كارلوس ف. بارباس في معهد سكريپس للأبحاث لتأسيس مكتبات أجسام مضادة مؤنسنة مخلقة، المبدأ الذي سجلت براءة اختراعه عام 1990 من قبل برايتلنگ وزملائه (براءة الاختراع CA 2035384)، وبالتالي يسمح بتخليق الأجسام المضادة المؤنسنة في المعمل من العناصر المتنوعة المخلقة.[24][25][26][27]
تعرض مكتبات الأجسام المضادة ملايين من الأجسام المضادة المختلفة على العاثية التي تستخدم عادة في صناعة الأدوية لعزل الأجسام المضادة العلاجية شديدة التخصص، من أجل تطويرها إلى أدوية أجسام مضادة وخاصة الأدوية المضادة للسرطان والالتهاب. ومن أنجح هذه الأدوية الأداليمومباب Adalimumab، الذي اكتشفته شركة كمبردج لتكنولوجيا الأجسام المضادة باسم D2E7 وقامت معامل أبوت بتطويره وتسويق. الأداليمومباب، جسم مضاد لـ TNF alpha، كان أول جسم مضاد مؤنسن بالكامل في العالم،[28] والذي حقق مبيعات سنوية تجاوزت البليون دولار.[29]
الپروتوكول العام
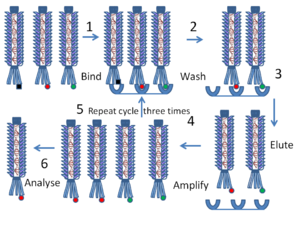
في ما يلي تسلسل الأحداث المتبعة في فحص عرض الآكلة لتحديد عديدات الپپتيد التي ترتبط بألفة عالية مع الپروتين المستهدف أو تسلسل الحمض النووي المرغوب:
- يتم تثبيت أو تجميد الپروتينات أو تسلسلات الحمض النووي المستهدفة على صفيحة معايرة دقيقة microtiter plate.
- يتم التعبير عن الكثير من التسلسلات الوراثية في مكتبة الآكلة على شكل اندماجات مع الپروتين المغلف للآكلة، بالتالي تكون معروضة على سطح الجسيم الڤيروسي. يتطابق الپروتين المعروض مع التسلسل الجيني داخل الآكلة.
- تضاف مكتبة عرض الآكلة هذه إلى الصحن ثم تُمنح الآكلة الوقت الكافي للارتباط، ثم يُغسل الصحن.
- تظل پروتينات عرض الآكلة التي تتفاعل مع الجزيئات المستهدفة ملتصقة بالصحن، بينما تنجرف الپروتينات الأخرى بعيداً.
- قد يتم شطف Elution الآكلة الملتصقة واستخدامها لخلق المزيد من الآكلات بإصابة العوائل الجرثومية المناسبة. تؤلف الآكلة الجديدة خليطاً غنياً، يحتوي على كمية كبيرة من الآكلات الغير مناسبة (أي الغير مرتبطة) والتي تكون موجودة في الخليط الأولي.
- الخطوة 3 إلى 5 تُكرر اختيارياً مرة أو أكثر، لإغناء مكتبة الآكلة بالمزيد من الپروتينات المرتبطة.
- يلي ذلك عملية تضخيم إضافية معتمدة على الجراثيم، يتم تتبع تسلسل الدنا الموجودة داخل الآكلة المتفاعلة لتحديد الپروتينات أو أجزاء الپروتينات المتفاعلة.
اختيار الپروتين المغلف
الآكلات الفتيلية
آكلات T7
موارد وأدوات المعلوماتية الحيوية
أصبحت أدوات قواعد البيانات والأدوات الحاسوبية لمحاكيات الحاتمة جزءاً هاماً من دراسة عرض الآكلة.[30] Databases,[31] وقد استخدمت البرامج وخوادم الوب بشكل موسع[32] لاستبعاد الپپتيدات غير ذات الصلة بالهدف،[33] تميز تفاعلات الجزيئات-الپروتين الصغيرة وخريطة تفاعلات الپروتين-پروتين. يمكن للمستخدمين استخدام البنية ثلاثية الأبعاد للپروتين والپپتيد المختار من تجربة عرض الآكلة لرسم خريطة للحواتم التمثيلية. توجد بعض الطرق الحاسوبية السريعة والفعالة أونلاين (على سبيل المثال EpiSearch [32] http://curie.utmb.edu/episearch.html ).
انظر أيضاً
تقنيات منافسة:
المصادر
- ^ أ ب Smith GP (June 1985). "Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface". Science. 228 (4705): 1315–7. Bibcode:1985Sci...228.1315S. doi:10.1126/science.4001944. PMID 4001944.
- ^ Smith GP, Petrenko VA (April 1997). "Phage Display". Chem. Rev. 97 (2): 391–410. doi:10.1021/cr960065d. PMID 11848876.
- ^ Kehoe JW, Kay BK (November 2005). "Filamentous phage display in the new millennium". Chem. Rev. 105 (11): 4056–72. doi:10.1021/cr000261r. PMID 16277371.
- ^ Malys N, Chang DY, Baumann RG, Xie D, Black LW (2002). "A bipartite bacteriophage T4 SOC and HOC randomized peptide display library: detection and analysis of phage T4 terminase (gp17) and late sigma factor (gp55) interaction". J Mol Biol. 319 (2): 289–304. doi:10.1016/S0022-2836(02)00298-X. PMID 12051907.
- ^ {{{1}}} patent {{{2}}}
- ^ "The Nobel Prize in Chemistry 2018". NobelPrize.org (in الإنجليزية الأمريكية). Retrieved 2018-10-03.
- ^ Explanation of "Protein interaction mapping" from The Wellcome Trust
- ^ Lunder M, Bratkovic T, Doljak B, Kreft S, Urleb U, Strukelj B, Plazar N (November 2005). "Comparison of bacterial and phage display peptide libraries in search of target-binding motif". Appl. Biochem. Biotechnol. 127 (2): 125–31. doi:10.1385/ABAB:127:2:125. PMID 16258189.
- ^ Bratkovic T, Lunder M, Popovic T, Kreft S, Turk B, Strukelj B, Urleb U (July 2005). "Affinity selection to papain yields potent peptide inhibitors of cathepsins L, B, H, and K". Biochem. Biophys. Res. Commun. 332 (3): 897–903. doi:10.1016/j.bbrc.2005.05.028. PMID 15913550.
- ^ Lunder M, Bratkovic T, Kreft S, Strukelj B (July 2005). "Peptide inhibitor of pancreatic lipase selected by phage display using different elution strategies". J. Lipid Res. 46 (7): 1512–6. doi:10.1194/jlr.M500048-JLR200. PMID 15863836.
- ^ Hufton SE, Moerkerk PT, Meulemans EV, de Bruïne A, Arends JW, Hoogenboom HR (December 1999). "Phage display of cDNA repertoires: the pVI display system and its applications for the selection of immunogenic ligands". J. Immunol. Methods. 231 (1–2): 39–51. doi:10.1016/S0022-1759(99)00139-8. PMID 10648926.
- ^ Gommans WM, Haisma HJ, Rots MG (December 2005). "Engineering zinc finger protein transcription factors: the therapeutic relevance of switching endogenous gene expression on or off at command". J. Mol. Biol. 354 (3): 507–19. doi:10.1016/j.jmb.2005.06.082. PMID 16253273.
- ^ أ ب "CAR T Cells: Engineering Patients' Immune Cells to Treat Their Cancers". National Cancer Institute. Retrieved 9 February 2018.
- ^ Løset, Geir Åge; Berntzen, Gøril; Frigstad, Terje; Pollmann, Sylvie; Gunnarsen, Kristin S.; Sandlie, Inger (12 January 2015). "Phage Display Engineered T Cell Receptors as Tools for the Study of Tumor Peptide–MHC Interactions". Frontiers in Oncology. 4 (378). doi:10.3389/fonc.2014.00378. Retrieved 9 February 2018.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ McCafferty, J.; Griffiths, A.; Winter, G.; Chiswell, D. (1990). "Phage antibodies: filamentous phage displaying antibody variable domains". Nature. 348 (6301): 552–554. Bibcode:1990Natur.348..552M. doi:10.1038/348552a0. PMID 2247164.
- ^ Scott JS, Barbas CF III, Burton, DA (2001). Phage Display: A Laboratory Manual. Plainview, N.Y: Cold Spring Harbor Laboratory Press. ISBN 0-87969-740-7.
{{cite book}}: CS1 maint: multiple names: authors list (link) - ^ Breitling, F.; Dübel, S.; Seehaus, T.; Klewinghaus, I.; Little, M. (1991). "A surface expression vector for antibody screening". Gene. 104 (2): 147–53. doi:10.1016/0378-1119(91)90244-6. PMID 1916287.
- ^ Barbas CF, Kang AS, Lerner RA, Benkovic SJ (September 1991). "Assembly of combinatorial antibody libraries on phage surfaces: the gene III site". Proc. Natl. Acad. Sci. U.S.A. 88 (18): 7978–82. Bibcode:1991PNAS...88.7978B. doi:10.1073/pnas.88.18.7978. PMC 52428. PMID 1896445.
- ^ Burton DR, Barbas CF, Persson MA, Koenig S, Chanock RM, Lerner RA (November 1991). "A large array of human monoclonal antibodies to type 1 human immunodeficiency virus from combinatorial libraries of asymptomatic seropositive individuals". Proc. Natl. Acad. Sci. U.S.A. 88 (22): 10134–7. Bibcode:1991PNAS...8810134B. doi:10.1073/pnas.88.22.10134. PMC 52882. PMID 1719545.
- ^ Barbas CF, Björling E, Chiodi F, Dunlop N, Cababa D, Jones TM, Zebedee SL, Persson MA, Nara PL, Norrby E (October 1992). "Recombinant human Fab fragments neutralize human type 1 immunodeficiency virus in vitro". Proc. Natl. Acad. Sci. U.S.A. 89 (19): 9339–43. Bibcode:1992PNAS...89.9339B. doi:10.1073/pnas.89.19.9339. PMC 50122. PMID 1384050.
- ^ Burton DR, Pyati J, Koduri R, Sharp SJ, Thornton GB, Parren PW, Sawyer LS, Hendry RM, Dunlop N, Nara PL (November 1994). "Efficient neutralization of primary isolates of HIV-1 by a recombinant human monoclonal antibody". Science. 266 (5187): 1024–7. Bibcode:1994Sci...266.1024B. doi:10.1126/science.7973652. PMID 7973652.
- ^ Yang WP, Green K, Pinz-Sweeney S, Briones AT, Burton DR, Barbas CF (December 1995). "CDR walking mutagenesis for the affinity maturation of a potent human anti-HIV-1 antibody into the picomolar range". J. Mol. Biol. 254 (3): 392–403. doi:10.1006/jmbi.1995.0626. PMID 7490758.
- ^ Barbas CF, Hu D, Dunlop N, Sawyer L, Cababa D, Hendry RM, Nara PL, Burton DR (April 1994). "In vitro evolution of a neutralizing human antibody to human immunodeficiency virus type 1 to enhance affinity and broaden strain cross-reactivity". Proc. Natl. Acad. Sci. U.S.A. 91 (9): 3809–13. Bibcode:1994PNAS...91.3809B. doi:10.1073/pnas.91.9.3809. PMC 43671. PMID 8170992.
- ^ Barbas CF, Bain JD, Hoekstra DM, Lerner RA (May 1992). "Semisynthetic combinatorial antibody libraries: a chemical solution to the diversity problem". Proc. Natl. Acad. Sci. U.S.A. 89 (10): 4457–61. Bibcode:1992PNAS...89.4457B. doi:10.1073/pnas.89.10.4457. PMC 49101. PMID 1584777.
- ^ Barbas CF, Languino LR, Smith JW (November 1993). "High-affinity self-reactive human antibodies by design and selection: targeting the integrin ligand binding site". Proc. Natl. Acad. Sci. U.S.A. 90 (21): 10003–7. Bibcode:1993PNAS...9010003B. doi:10.1073/pnas.90.21.10003. PMC 47701. PMID 7694276.
- ^ Barbas CF, Wagner J (October 1995). "Synthetic Human Antibodies: Selecting and Evolving Functional Proteins". Methods. 8 (2): 94–103. doi:10.1006/meth.1995.9997.
- ^ Barbas CF (August 1995). "Synthetic human antibodies". Nat. Med. 1 (8): 837–9. doi:10.1038/nm0895-837. PMID 7585190.
- ^ Lawrence S (April 2007). "Billion dollar babies--biotech drugs as blockbusters". Nat. Biotechnol. 25 (4): 380–2. doi:10.1038/nbt0407-380. PMID 17420735.
- ^ Cambridge Antibody: Sales update | Company Announcements | Telegraph
- ^ Huang J, Ru B, Dai P (2011). "Bioinformatics resources and tools for phage display". Molecules. 16 (1): 694–709. doi:10.3390/molecules16010694. PMID 21245805.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - ^ Huang J, Ru B, Zhu P, Nie F, Yang J, Wang X, Dai P, Lin H, Guo FB, Rao N (January 2012). "MimoDB 2.0: a mimotope database and beyond". Nucleic Acids Res. 40 (Database issue): D271–7. doi:10.1093/nar/gkr922. PMC 3245166. PMID 22053087.
- ^ أ ب Negi SS, Braun W (2009). "Automated Detection of Conformational Epitopes Using Phage Display Peptide Sequences". Bioinform Biol Insights. 3: 71–81. PMC 2808184. PMID 20140073.
- ^ Huang J, Ru B, Li S, Lin H, Guo FB (2010). "SAROTUP: scanner and reporter of target-unrelated peptides". J. Biomed. Biotechnol. 2010: 101932. doi:10.1155/2010/101932. PMC 2842971. PMID 20339521.
{{cite journal}}: CS1 maint: unflagged free DOI (link)
قراءات إضافية
- Ledsgaard L, Kilstrup M, Karatt-Vellatt A, McCafferty J, Laustsen AH (2018). "Basics of antibody phage display technology". Toxins. doi:10.3390/toxins10060236. PMID 29890762.
{{cite journal}}: CS1 maint: unflagged free DOI (link) - Selection Versus Design in Chemical Engineering
- The ETH-2 human antibody phage library
- Sidhu SS, Lowman HB, Cunningham BC, Wells JA (2000). "Phage display for selection of novel binding peptides". Meth. Enzymol. Methods in Enzymology. 328: 333–63. doi:10.1016/S0076-6879(00)28406-1. ISBN 9780121822293. PMID 11075354.
وصلات خارجية
| مراجع مكتبية عن عرض الآكلة |